周报_2022_1.10-2022_1.15_杨延青
2022.1.10-2022.1.15工作汇报
一、共价晶体结构数据库构建及共价模型构建
1. 背景
已有的共价数据库:
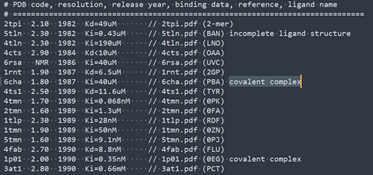
(1)共价数据库收集了所有可能形成共价的化合物的相关信息。一共有10327条数据(化合物SMILES和蛋白Uniport对)。但是其中的晶体结构记录只有340条,并且没有给出PDB ID。

(2)该数据库和CovalentInDB非常相似,当前网站已出现错误,无法正常访问。

(3)PDBbind有晶体结构,有活性信息,涉及到共价信息的有381条。
小结:
由以上3个数据库可知,目前仍未有完整的共价晶体结构数据库。
所以对PDB数据库进行清洗获取共价晶体结构。
并以共价数据库为基础建立深度学习模型对共价化合物活性进行预测。
(4)共价类型
1. 与半胱氨酸(CYS)结合的共价抑制剂
2. 与丝氨酸(SER)、酪氨酸(TYR)和苏氨酸(THR)结合的共价抑制剂
3. 与赖氨酸(LYS)结合的共价抑制剂
4. 与谷氨酸(GLU)和天冬氨酸(ASP)结合的共价抑制剂

2. 方法
(1) 下载完整的PDB晶体结构数据库
(2) 筛选出PDB数据库中有LINK信息的pdb文件
(3) 根据筛选出的pdb文件中的LINK信息对共价pdb结构进行分析
(4) 根据LINK进行筛选:
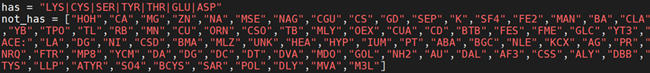
对于每个pdb的LINK信息,首先遍历LINK中每一行,如果有行内含有以下氨基酸的同时,
又没有金属离子等的非配体化合物。则将pdb保留下来,即可能的共价结构。筛选之后,
从pdb晶体库中筛选出了6349个晶体结构。但这些结构中并不都是共价的。

(5) 对6349个晶体结构的LINK信息中的残基进行统计,统计结果如下:
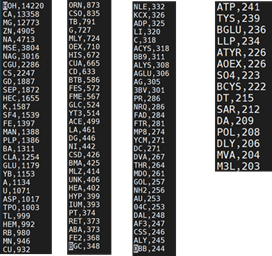
(6) 对(5)中的残基进行查看,找出不需要的包括糖等在内的残基名字,以同样的方式对6349个结构再次筛选,这次又从6349个pdb结构中筛选出了5049结构。
(7) 结合pymol和PDB文件中的LINK信息对剩下的5049个结构再次进行筛选。最终选出3363个晶体结构。
(8) 为了防止下图中的结构,对蛋白氨基酸残基数量进行限制,去掉残基数量小于等于20的ID。
3MBS:(×)

(9) 经过统计残基数小于等于20的结构一共有46个
(10) 去掉残基种类小于等于10的ID。
(11) 最终一共剩余3300个结构。
3. 结果
3.1 数据库结果准确度查询
随机抽样进行数据库准确度验证,即随机从3300个结构中抽取10个进行肉眼观察,看有多少个是共价结合的结构。
结果显示随机抽取的10个结构全部都是共价结构。
3.2 模型建立
(1) 先将PDBbind中的共价结构以及标签取出来组成数据库,建立初步模型。
(2) 模型结构如下图所示:
提取整个配体周围的残基信息
提取配体每个原子周围的残基信息
共价信息等构建深度学习模型预测活性。
二、ALKBH5
1. 活性结构
2. 将Compound2对接到ALKBH5和FTO口袋位置的所有SER丝氨酸。
(1)首先找出ALKBH5和FTO口袋周围的所有丝氨酸。如下所示:
ALKBH5:189,217,219,224
FTO:318,240,246
(2)针对上述ALKBH5和FTO口袋周围的所有丝氨酸,使用上述Compound2进行共价对接,对接结果见:Covalent_Docking.pse
3. 结构改造一
将Compound2与之前虚拟筛选有活性的化合物进行结构比对和结构拼接。
根据建议,不断的延长甲氧基的侧链。一共设计了上述10个化合物。并将此10个化合物与ALKBH5的217位丝氨酸进行共价对接,对接结果见设计的结构共价对接结果.pse
4. 结构改造二
将Compound2与之前虚拟筛选打分最高的化合物进行结构比对和结构拼接。
对接结果见设计的结构共价对接结果11-15.pse
