Structure&Pipeline
研究背景:表型的差异对应实体不一定就是致病的核心,即靶点。因此才会有富集分析扩大范围找到真正的致病点。但是尽管这一过程很好实现,但是由于数据的缺省、异质性、批次效应等等等,能够进行的分析工作十分局限。因此,近些年多组学整合成为一个热点,旨在利用有效的算法弥补数据上的不足,以顺利推进下游的分析。本研究提出了一种通过可逆模型捕捉组学实体之间依赖关系,进行多组学整合与靶点发现的方法。
核心思路解读:1)如果是自建数据集,大概率可以进行差异表达分析,则可以投入表达数据,做差异表达分析(当然也可以做其他的差异分析、突变分析等等等,关键是得到初始的焦点实体)。如果做不了,那就从输入或者任意实体开始,然后进行下一步分析。
2)有了焦点实体,我们还需要候选实体,这些候选实体是可以从焦点实体中富集的实体集,也可以是全部的数据库内的实体。如果有富集的过程,范围本身就比较小,速度会快很多。如果没有富集,那范围比较大,速度会慢很多很多。我们算作快速模式和精准模式。然后进行下一步。
3)根据任务的定义的所有实体,调用数据——————>数据的组织形式还是个大问题。
4)TVM优化指标量(目前的指标量:微分系数,Spearman相关系数),筛选出相关性最大实体,形成图------------------------------>图生成过程是否合理呢?病患图生成和正常样本的图生成分开,最终对比生成的图,交集点即为关键的靶点,在病患途中到焦点实体的传播路径即为可能的病理机制变化。
5)临床相关性,利用生成的图进行下游的病患的生存预测,理论上能得到一个高的一致性与cox评估。
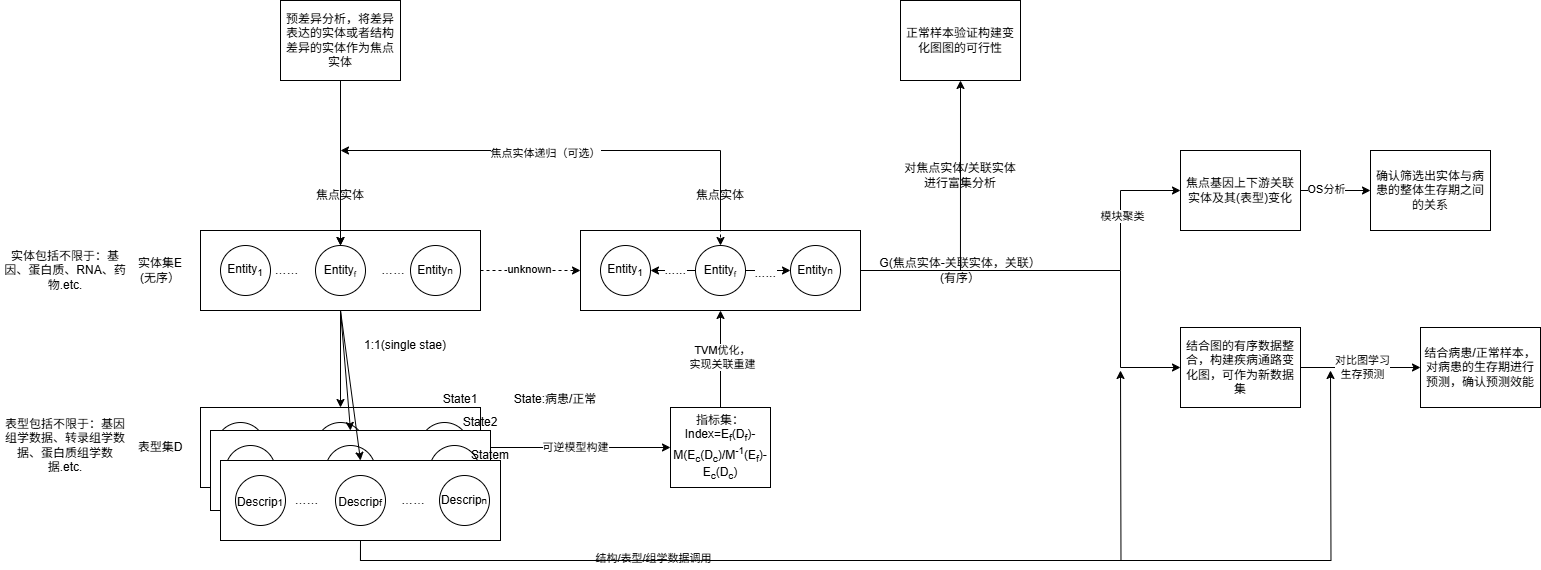
设计原则:1)能用的样本全用上,只考虑分子标签集的变化。2)轻量化传参
重点及难点:1)病变机制究竟是态的变化还是路的变化?2)路径生成
管线设计:1)数据库设计:详见Multi-omics数据库设计
存在问题:数据的组织形式
组学数据的组织形式:
对单个基因G,可用的量化数据如下:
1.Copy Number Segment(log2比值离散化)-2(纯合缺失), -1(杂合缺失), 0(中性), +1(低扩增), +2(高扩增)
2.Differential Gene Expression{ -1, 0, +1 } 对应下调/无/上调
3.Gene Expression Quantification(log2(TPM+1))0(无表达)→ 一般在10以内(如MYC≈6.5)
4.Masked Somatic Mutation突变频谱计数,整数计数(0~N)
5.Methylation Beta Value:原始β值,比例型(0~1)<0.3为低甲基化,>0.7为高甲基化
6.miRNA Expression Quantification,log2(RPM+1),0(未检测)→8(高表达miR-21)
7.Protein Expression Quantification,Z-score归一化,标准化连续值,(原始值-平均值)/标准差,典型范围-3~+3
8.Slide Image & Tissue Microarray Image,特征提取值
1)二维张量/矩阵形式
column=(case_id,genes×omics)
对基因组就需要做笛卡尔积了
图生成过程是否合理呢?
dumbass模式:
逻辑:取三个癌基因,占比1,掺进去一个,那么就是大于等于三分之二。不妨做一个二分类,大于等于1和大于等于三分之二。包含不要紧,就看有没有掺进来但是还是被划分到大于1的情况,那这个肯定是癌相关基因。当时又怎么从数值上定义这种包含的标签呢,比如我们选择一个参考点5,5-1=4,5-2/3=13/3.或者说标签变量能不能是个范围,那肯定是个能(p,1),(p,2/3)
模型首选是:vae,resnet&xgb
输出:(p,1),(p,2/3),令p=1,当然三可以换成的别的5啥的。虽然但是,这个不能表示包含关系。怎么才能有表示包含关系的数/数据?向量之间是存在包含关系的吧,
可以两个空间的特征向量为D=(p,1)和d=(q,2/3),然后D包含于d,把这种包含关系定义为:D//d,且|D|<|d|.可以求出,复数域了到……不太行。
在策略训练策略上,两阶段训练,一阶段:全部用癌基因训练,预测变量都是1,出来一个模型。然后引入一个未知基因1或者2/3,训练,再用原来的1来验证,哪个准确度高哪个就是保留哪个模型,并且标记这个基因为癌基因/非癌症基因。总感觉就那样,不太行。还是选用成药的靶点。
明确思路:两个阶段训练:1)利用已知癌基因(5个)的组学预测目标基因中癌基因比例(因为全是癌基因阳性,标签量(比例)全为1)2)向目标基因中掺入3个未知基因(假设标签量(比例)为2/5),微调训练,由癌基因的数据进行验证。引入评价掺入基因的癌症影响的指数:CII=1-ratepre,可见CII->0,掺入基因对癌症的越关键。
这么一整的话,rf/xgb好像不太行,微调不要。resnet,vae都可以
补充说明:
CounterPaper/Model:GeSubNet( GESUBNET: GENE INTERACTION INFERENCE FOR DISEASE SUBTYPE NETWORK GENERATION)
