CovalentDock 共价对接软件
CovalentDock介绍
--Edited by 石禹龙 2021-12-21
CovalentDock是2013年发表在JCC上的基于AutoDock开发的免费开源共价对接软件。
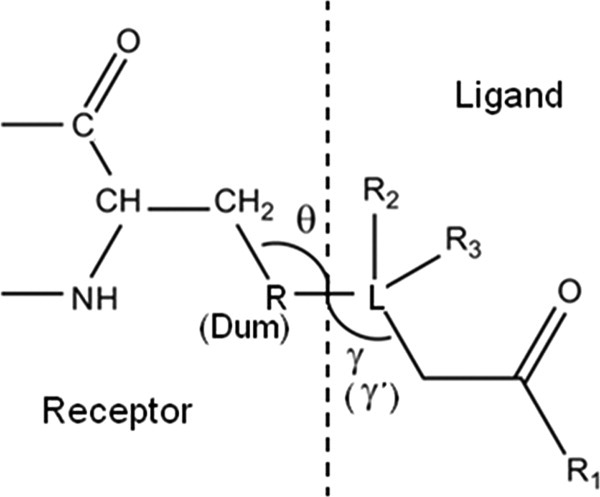
特点:
- 自动化程度高,可自动识别蛋白和配体的共价结合位点,适合虚拟筛选;
- 考虑了共价结合的能量贡献(Gaussian),打分数值较高(Maestro CovDock共价打分较低)
缺点:
- 目前能自动识别的共价反应类型较少,只支持SER、CYS;
- 软件许久没有更新,印象中需要用到老版本的amber12,如果要用较新的amber,需要根据amber版本修改源代码,比如tleap的处理;
- 后处理比较麻烦。
- 只能识别特定的反应基团(见原始文献)
使用感受:
- 虽然能自动,但是有时生成的构象没有形成共价键,这类构象往往打分会差很多,容易辨识;
- 生成的中间文件比较大,要及时清理。
此处根据使用情况,以amber 16为例,介绍CovalentDock的使用步骤,修改后的CovalentDock源代码见附件,22202服务器(172.21.85.12)上已配置好环境,可正常使用。
使用步骤 命名蛋白为test_receptor.pdb,小分子为test_ligand.mol2,对接坐标中心为center。
- $CD_HOME/preprocessing test
- cd bond$CD_HOME/covalentGrid -p test_bond.gpf -l test_bond.glg
一般来说,共价结合会生成两种手性构象,需要都保留。
- $CD_HOME/covalentDock -p test_ligand_bond_1_C17_1_mini.dpf -l test_ligand_bond_1_C17_1_mini.dlg
- $CD_HOME/covalentDock -p test_ligand_bond_1_C17_2_mini.dpf -l test_ligand_bond_1_C17_2_mini.dlg
- grep '^DOCKED' test_ligand_bond_1_C17_1_mini.dlg|sed 's/DOCKED: //g' >
- test_ligand_bond_1_C17_1_mini.pdbqtgrep '^DOCKED' test_ligand_bond_1_C17_2_mini.dlg|sed 's/DOCKED: //g' > test_ligand_bond_1_C17_2_mini.pdbqt
- # Covalent Score
- grep 'Estimated Free Energy of Binding' test_ligand_bond_1_C17_1_mini.pdbqt test_ligand_bond_1_C17_2_mini.pdbqt|sort -t '=' -nk 2
另外,对接过程中产生的虚原子需要替换为原来的原子类型。
- # 例如CYS
- sed -i 's/V1/C/g' test_ligand_bond_1_C17_1_mini.pdbqt
