log
2025-7-4:75.2~/Backup/History_on_Pocket2Druggability是该课题的备份路径。
2025-7-7:等明天讨论的结果
2025-7-8:现在的结果是需要完成的一个管线:
MD数据->fpocket/d3pocket->比较结果,发现其中口袋的特征,统计规律
2025-7-14:有点搁置了,感觉锦添师姐数据应该差不多了,稍微花点时间部署D3pocket & fpocket
尽快提交下。描述一下数据集:目标文件夹,75.2~/jintian/AI-NCI/
dynamicPDB:原始数据,每个体系100ns&1000帧
MISATO:另一个数据集,每个体系10ns&100帧
dynamicPDB_2050710_clean是dynamicPDB处理过的数据
注:每个体系文件中的.dat文件是RMSD的记录文件,注意clean中是参考的原始蛋白结构,MISATO参考的是初始结构。另外MISATO中的体系是带配体的复合物体系,MISATO目录下的apoprotein就是没有配体的了。另外,MISATO的体系存在多链的情况,存在蛋白链的冗余。
复制到 我的Backup/Dataset
2025-7-16:虽然还没cp完,但是可以先部署d3pocket,需要考虑哪台服务器,1)空间足够 2)cpu性能够。不用移植,85.23上面就有,虽然但是这文件组织真的给我整吐了,强迫症犯了。真的好想全部删了重写,哪有这么写代码的,后期维护起来的想想头皮就发麻。算了,算了,这样吧
测试:85.23:/software/D3Pockets_WEB_UCB
conda activate D3pockets
nohup bash D3Pockets_MD_Script.sh &
pid:201156
测试完成,看看啥样。
mad,这都输出了啥啊,样本量不要太大了吧。。
2025-7-17:需要整理下,最终数据的数据的类型,对应说明的问题。
2025-7-21:活人微死,谢谢。真的,这周开始D3pocket的数据分析了。统计个啥呢。基于什么呢,基于形状吗?考虑残基吗?还是啥啊,好问题。先做出一版来吧
2025-7-22:嘿,nc的一天从忘记带电脑充电器开始。虽然但是想一下任务:路径整理到数据库中,后续直接跨服务器操作。先录入数据库。
数据库的建立
R==relation,E==entity
E<-Md_files(filename,filepath,filetype)好像只需要这么一个简单的表就可以了。数据库就命名为Files,还是没有还是,没必要。起名字啥的最讨厌了,FILEs太笼统了,好不容易起了个DataFiles;
有些需要注意的:1)路径 2)更新
接下来就是开始,还好写了log,不然文件组成就要了老命了。根据需求建表。需求的是蛋白pdb文件和轨迹xtc文件。现在还需要稍微转换下:
1)gmx转换
2)python转换(MDanalysis)
我的建议是都来,然后比较下。
现在先用python转换:
/home/databank/gxxu/Backup/Moleculardynamics/4D3pocket_input/shs$ python 2xtc.py -i ../../Source/dynamicPDB_250710_clean/ -o ../
修改一下d3pocket的脚本,改成每个对一个。
2025-7-23:部署在75.4,conda,pymol==2.2.0为啥安装不上。哎,其实自己不太想面对,这python的版本太低了。一更新,tmd就有可能要动底层代码。就很难受。为什么啊,为啥是2.x版。要不这样,运行下脚本,然后一个个统计,稍微写了一个D3Pocket3(75.4)
2025-7-24:pdbdynamics已经转换为d3pockets的输入文件了。然后MISATO换分为单链也处理完了,但是脚本是错的。注释一下,4D3pocket_input中MISATO都没有单链的
现在把pdbdynamics的信息录入数据库。后面把MISATO处理完蛋白链冗余后也录入。
2to3 -w D3Pockets_WEB_UCB给升级到了python3了,但是出现了不兼容问题,
bug记录:
d3pocket.py中96行,缩进
drawnetworksinmoregraph.py 200行,括号封闭
356行,输出语句
clusterpkt_resid.py", line 14,废弃函数sys' has no attribute 'setdefaultencoding'.
drawnetworksin1graph.py", line 185,prenode = sorted(confpkt.items(), key=lambda item: item[1], reverse=True)[0][0]
在 Biopython 1.78+ 版本中,SCOPData 模块已被弃用并移除。
protein_letters_3to1 函数已迁移到 Bio.Data.IUPACData 模块。所以修改draw_sitestable.py文件。
现在最大的问题是MISATO,好象处理完了//NOOOO,没有提取,但是都是默认A链。路径:/home/databank/gxxu/Backup/Moleculardynamics/Source/MISATO/MISATO_MD_pdbs_apoprotein_mono,nohup python batch_extract.py --input ../../MISATO_MD_pdbs_apoprotein --output ../ &
您猜怎么着,还是不行。
2025-7-25:为啥,升级到python3,这结果就天差地别了。我再相信一次,怀疑是重构的时候的问题,
bug:
文件: genpktsmvbyres.py
行号: 164
错误内容: raise "error no 1"
文件: ressite_stable.py
行号: 57
错误内容: raise "error!!"
原因: Python 3不再支持直接使用字符串作为异常对象(这是Python 2的特性)
文件: drawnetworksinmoregraph.py
错误类型: ParseError
细节: 在201行20列附近遇到意外的冒号(:),就是上面括号的问题
文件: format_pocket.py
错误类型: ParseError
细节: 在19行7列附近遇到意外的换行符(\n)
drawnetworksinmoregraph.py", line 355
print "finsh: ",netinf
mmd,还得手动来。代码写得稀烂,2to3根本跑不动。开始修bug,
read_traj_single_frames.py输出问题
PyPocket_Detect4MPI.py输出问题
喵的,直接全部修了吧。现在有一版了,还是不行
2025-7-26:数据准备好了,就看如何升级或者用旧版.又重新开始了
妥协以下,D3pocket2-对应原来代码,妥协不了一点,pymol都装不了。
D3pockets对应可能转换后的代码。
2025-7-31:那个问题不是问题
75.4上的测试是2to3,然后手动修改的代码。(D3pocket3..)
2025-8-1:开始改bug,真不如重写
read_traj_single_frams:for id in outrajid: # ❌ id 是内置函数 id()
一轮修改,先整合到一起了
二轮修改,逻辑重构
三轮修改,bug修改
2025-8-16:85.23上批量处理获取的脚本已经完成在dddc/gxxu/D3*/D3*/D3Pockets_MD_Batch.sh
应该没啥问题,测试pid:75271。另外利用ai检查了detect相关的代码。
2025-8-18:一步步来,现在我们专注于输入输出流,不注重于算法。并且对每个模块进行单项测试测试的路径在75.4/home/dddc/gxxu/D3pockets/test,环境D3pockets
测试命令:python main.py -p ./data/look.pdb -x ./data/md-nowater-every1ns.xtc
今天测试到,pdbfiles=pocket_detector.cluster(output_dir=os.path.join(args.output,'clustered'),cluster_method=args.cluster_method,write=True)
1/29测试通过
差不多可以进行到detect的测试:2/29
开始测试mpi下的detect,测试路径:75.4/home/dddc/gxxu/test/test_2_29
测试命令:mpiexec -n 4 python main.py -p ./data/look.pdb -x ./data/md-nowater-every1ns.xtc
暂时没通过,在修改分布式问题。仔细分析下来,pocket_detect类并不合理。不如单纯的函数实现...
函数级隔离过不去了。
391197,测试一下会不会结束。感觉不会,这样吧,我们先跳过并行部分,专注于文件流的处理。明天继续2/29测试。
2025-8-19:2/29测试通过。并行化以及detect过程的优化后面再说,现在开始3/29
2025-8-20:3/29测试通过,大差不差,series.py好像没啥用。开始4/29测试:getpits,测试通过。
开始5/29测试:densityball_clu.py,测试通过。开始6/29测试:genpml.py。开始7/29测试:genstable_pml完成测试。开始8/29测试。
2025-8-21:8/29测试通过。开始9/29测试:summary,测试通过。下一个测试:10/29:genpocketsmvpml。测试通过。11/29:gennetwork1input测试通过。下一个12/29:drawnetwork
2025-8-22:草了,不是代码的问题。准确来说不是我修改后代码的问题。是tmd他默认第一个就是一个size=1的节点。问题出在前面没排序。12/29测试通过。13/29:gennetworksinput测试通过。
结果和原来的有细微的差别。
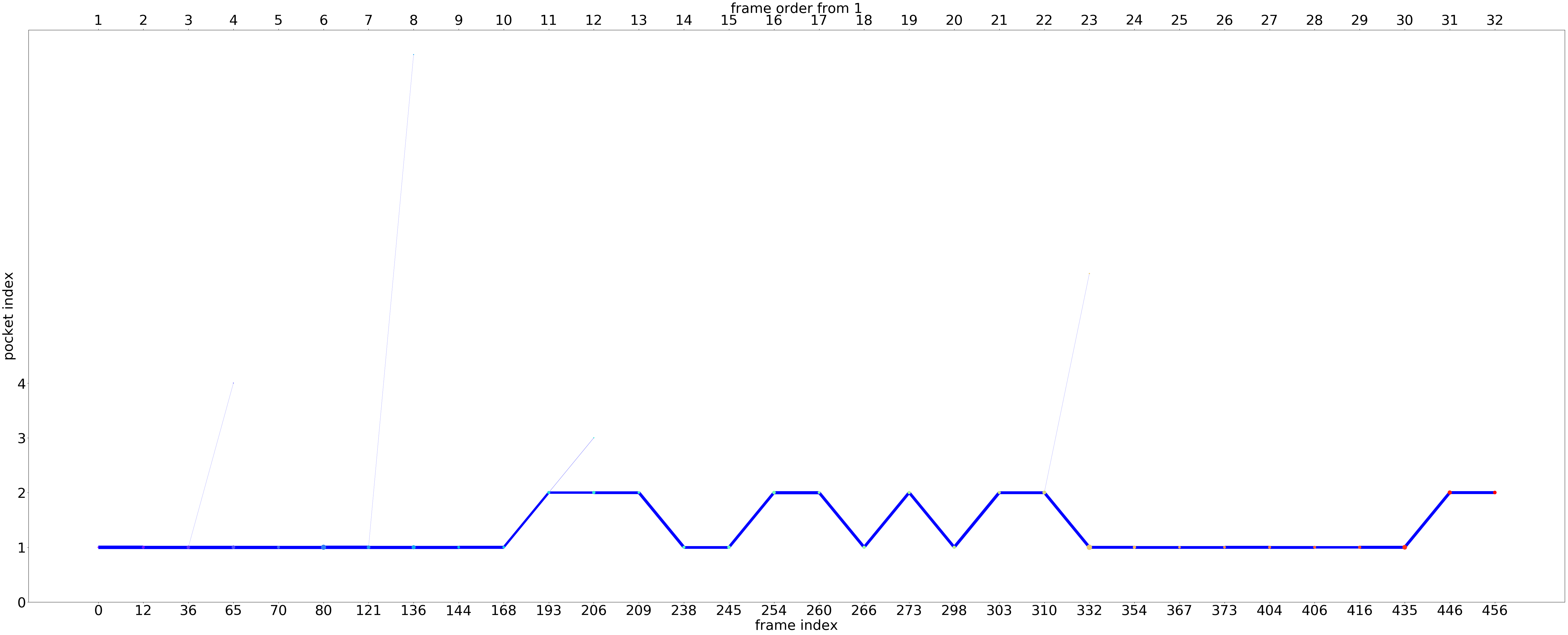
上面之前,下面之后。
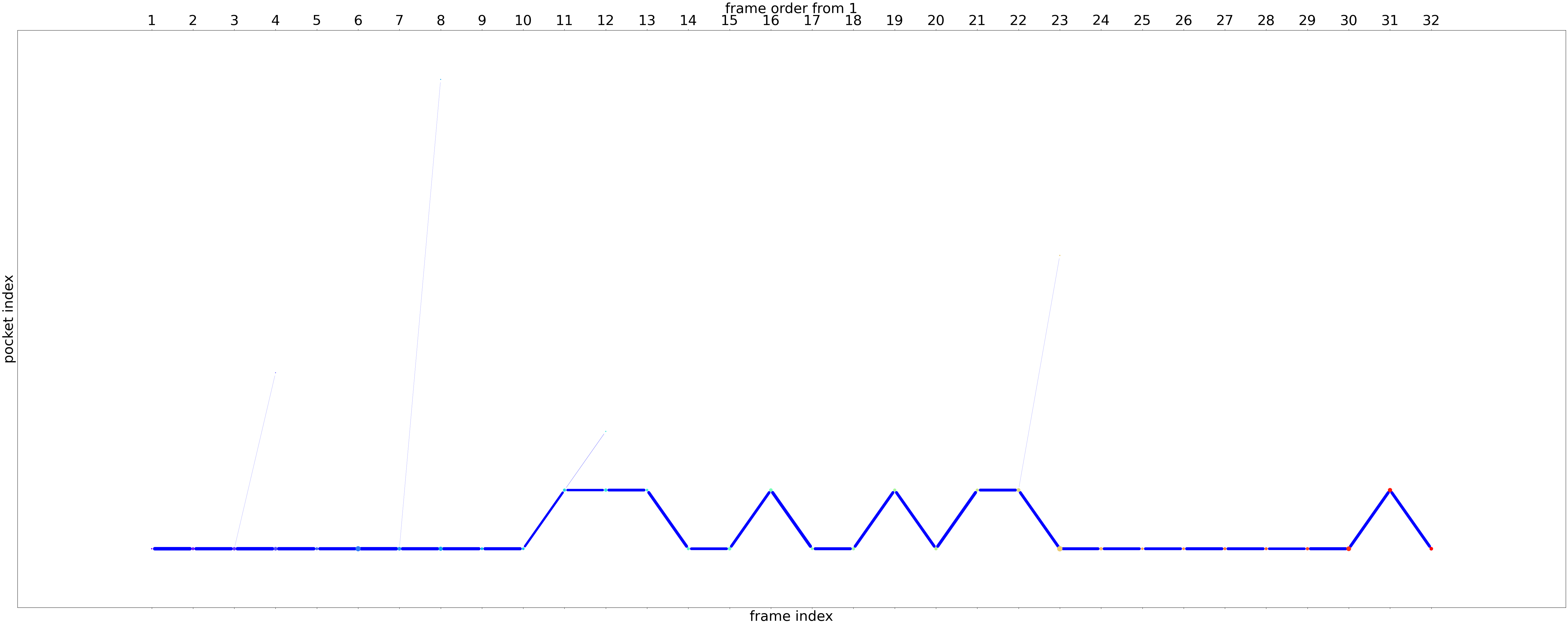
源自于聚类的结果。
开始14/29测试:drawnetworksinmoregraph.py。之前的报错就出在这里。删了,我们就用默认的。测试通过。开始15/29测试,drawnetworksin1graph.py。测试通过。16/19测试network2mvpml,17/29测试gencontinuity_pse.py通过。18/29测试:getatom,测试通过。
19/20:ressite_stable测试通过。20/29:draw_stable:测试通过。
2025-8-23:21/29测试:gen_colorpktresipml,测试通过。22/29测试:create_sqlite.py,改成mysql的,手动创建这么一个库。ok,75.2 库:pktsatmclu,表:CREATE TABLE IF NOT EXISTS pktsatmTab (
id INT PRIMARY KEY AUTO_INCREMENT,
cluid VARCHAR(255) NOT NULL,
clusize VARCHAR(255) NOT NULL,
pktatmfile TEXT,
simm_avg VARCHAR(255) NOT NULL,
pktatmfiles TEXT NOT NULL
) ENGINE=InnoDB DEFAULT CHARSET=utf8mb4;
一下内容遗弃了:SQLITE_DBNAME = "pktsatmclu.db"
SQLITE_TABLE = "pktsatmTab"
conn=sqlite3.connect(SQLITE_DBNAME)
cu=conn.cursor()
create_tb_sql='''CREATE TABLE if not exists pktsatmTab (
id integer primary key autoincrement,
cluid TEXT NOT NULL,
clusize TEXT NOT NULL,
pktatmfile TEXT ,
simm_avg TEXT NOT NULL,
pktatmfiles TEXT NOT NULL
)'''
try:
cu.execute(create_tb_sql)
except:
print ("Table exitst")
cu.close()
conn.commit()
conn.close()
下一个测试:23/29:clusterpkt_resid,测试通过。下一个:24/29:genpktsmvbyres,测试通过。下一个测试25/29:gencommonpktmvpml,测试通过。下一个测试 26/29:drawmat_heatmap
通过。死了得了。27/29:volume_correlation,测试通过。28/29:matrix2heatmap,测试通过。
29/29:selectPocketCorrelation,测试通过!!
2025-8-24:continuity那里用新的pymol打开还是有点问题。总之除了并行,所有测试结束了。接下来是优化,重整化,与新功能开始。现存档一下:75.2/home/databank/gxxu/Backup/History_on_D3pockets_2.0/D3pockets_py3
pdbdynamic计算中断:3mfd,重启了。
2025-8-25:开始D3pocket的优化,首先一点是绘图优化。matrix2heatmap用一个函数测试:测试通过。
2025-8-26:Drawer3D类统一生成Pse文件,跳过pml的生成,pml内容写入log文件。测试路径还是:/home/dddc/gxxu/testpy3_re/test_re_1 测试通过。
2025-8-27:continuity的pse文件也通过genPse函数,测试路径还是/home/dddc/gxxu/testpy3_re/test_re_1,草了,stability咋还不能用了,stability写错了。现在测试通过了。开始下一轮修改,test_re_2:drawnetworksinmoregraph.py,drawnetworksin1graph.py,drawnetwork.py 主要是优化整合这三个函数。不过首先,我把matrix2heatmap整合到drawer2D中了,没啥问题,但是漏掉了correlation的pse绘制过程,这必须整上,然后再drawnetwork,genCorrelationPse,测试路径:/home/dddc/gxxu/testpy3_re/test_re_2,测试通过。现在开始关于network的draw整合.
我稍微试一下drawmorenetwork替到drawnetwork的,结果是不是一样的。统一一下输入绘制network的文件的内容格式,第一列都不带路径。问题在于图生成的逻辑tmd统一,现在75.4中是1的生成逻辑,本地是s的生成逻辑。
2025-8-28:事实证明用s的生成逻辑就可以。然后整合到drawer2d上。drawnetwork*都整合到Drawer2D中了,测试通过,test_re_2结束
2025-8-29:今天,我么写一个GPU加速pocket_detect的版本,test_re_3先修改pocket_detect中调用的函数,然后再test_re_4中完成CUDA加速版本,混合版本之后再说。算了,哥们懒得优化这些函数来,直接写核函数哪
策略:编写C++/CUDA核函数 b. 创建Python调用封装 c. 优化数据传递 d. 性能测试
2025-8-29:对pdbdynamic已经去全部处理完了。1185个
2025-8-31:CUDA并行化,先到这里吧。在另一台电脑上。先处理pdbdynamics的结果,处理脚本在p75.2:~/dbdynamics/analysis,明天在继续分析
2025-9-1:分析的结果出来了,有个问题:为啥pdbid是上万个..
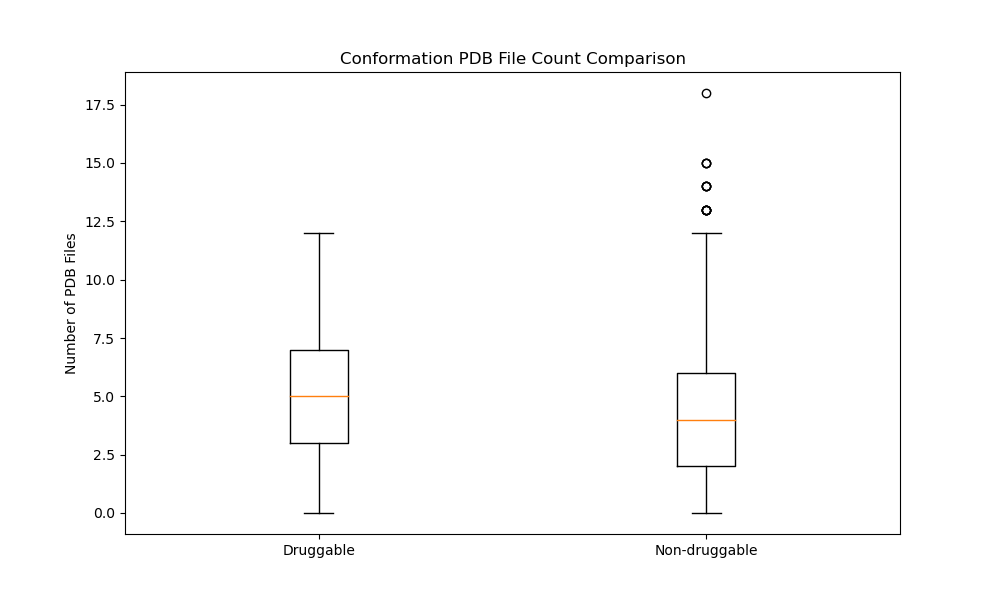
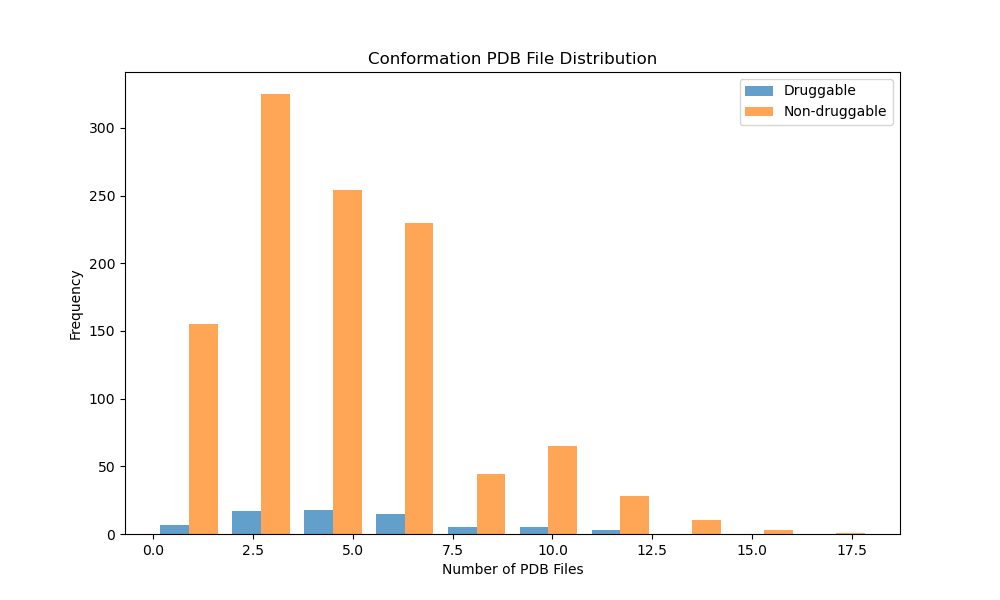
1185pdbid中只有13个成药的蛋白。以下是分析输出记录:
‘’‘从数据库中获取了16503个PDBID(去重前)
去重后剩余15373个唯一PDBID
Analyzing PDBIDs: 100%|██████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████████| 1185/1185 [10:03<00:00, 1.96pdbid/s]
处理的PDBID总数: 1185
其中成药蛋白: 13个
非成药蛋白: 1172个’‘’
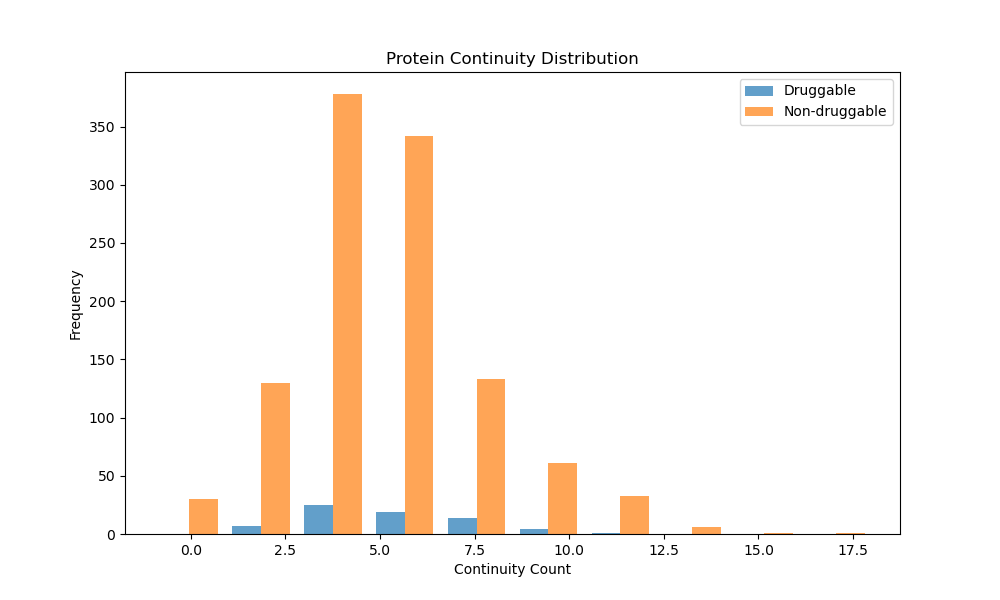
显然统计量太小,两者没有显著差异
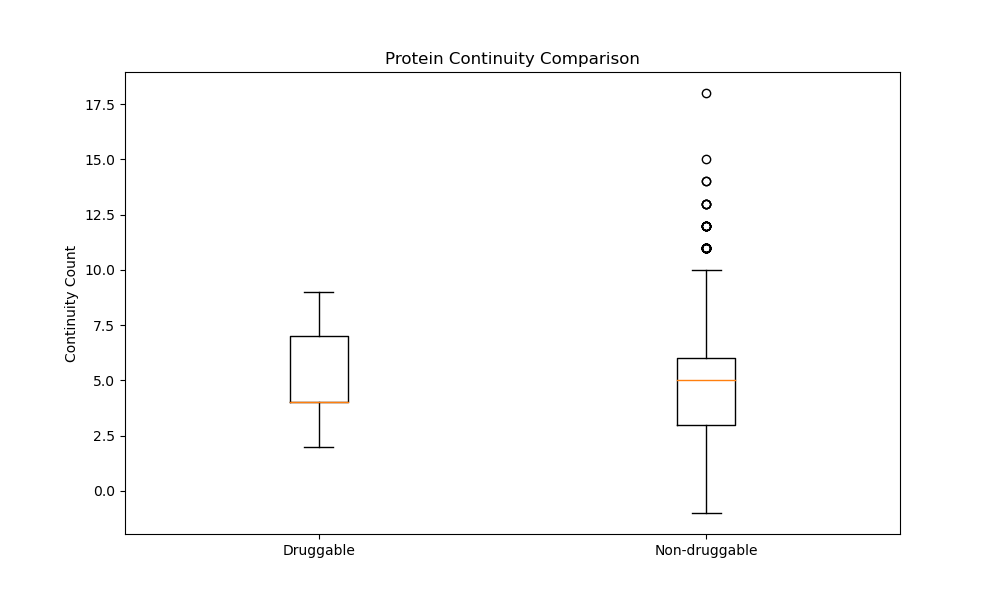
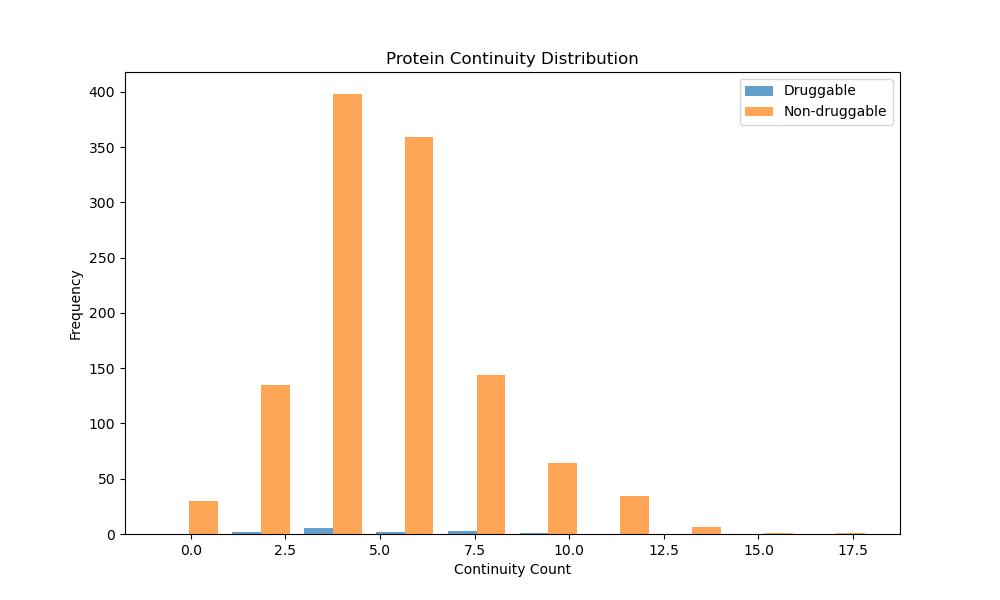
以上都是continuity,以下是stability
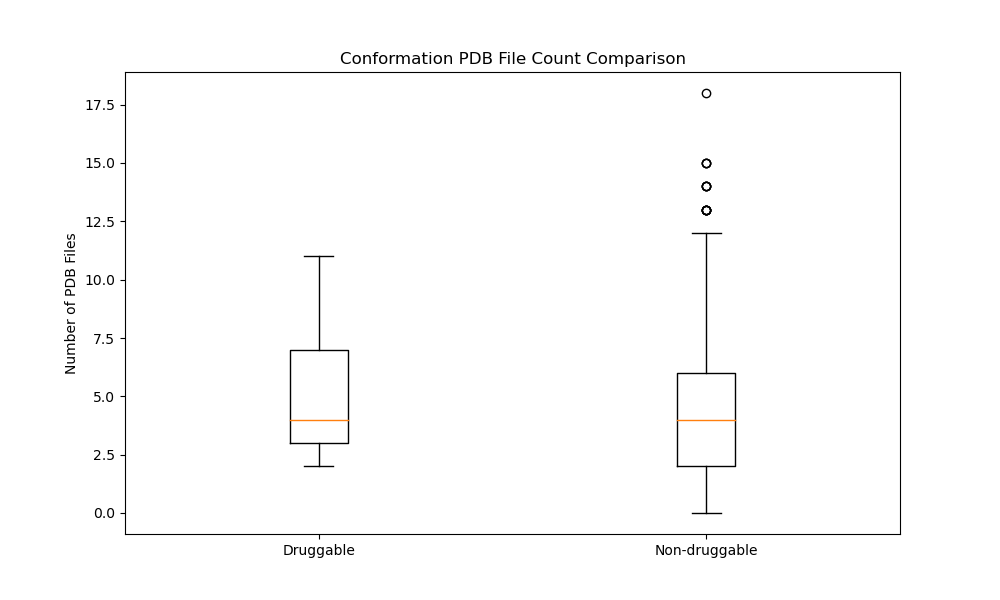
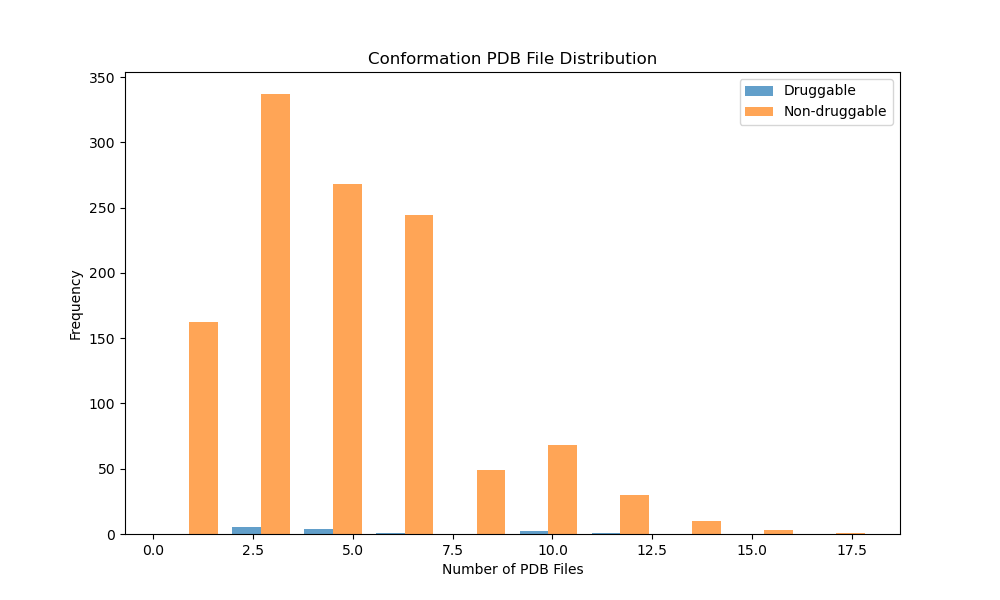
以下是correlations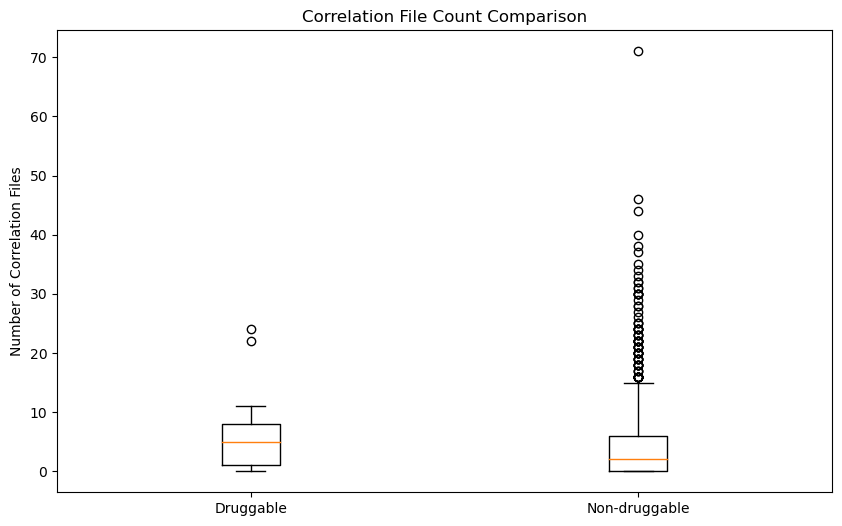
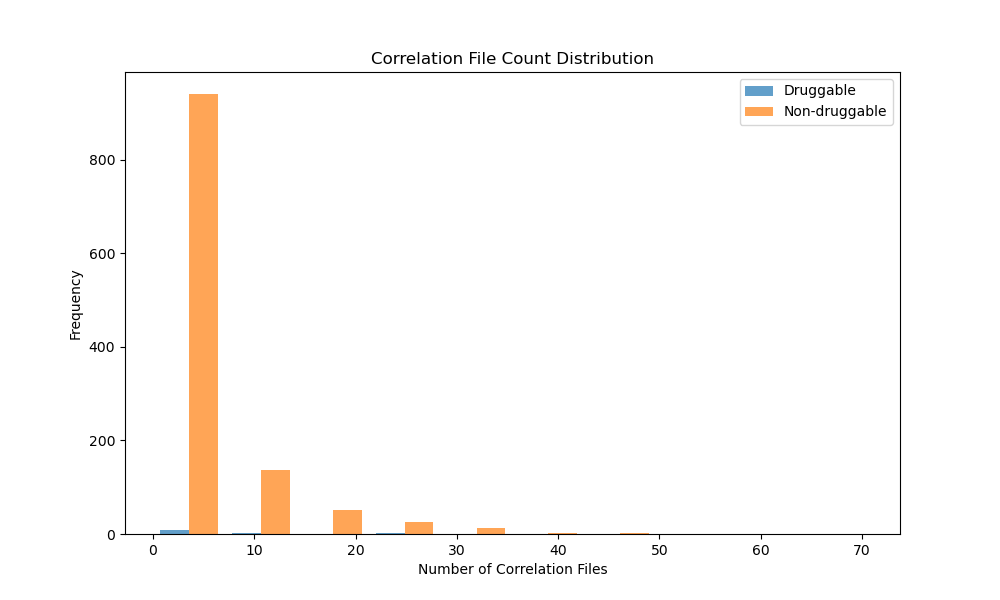


现在开始把MISATO_over 用import2mysql导入DataFiles数据库,然后用D3pocket*MD*Batch,处理,输出到/home/databank/gxxu/D3pocket_Result/MISATO
85.23:~/gxxu/D3P~WEB/**/read_traj* .py 增加了帧文件的导入方式,然后修改了批处理过程。
DataFiles增加帧文件表。
有点问题,gene_target中是肿瘤的,我们还是要所有的,所以新建了FDA_approved表。新的结果替换了原来的结果。还是在analysis中,本地是(analysis_drugbank)
新的结果如下:
continuity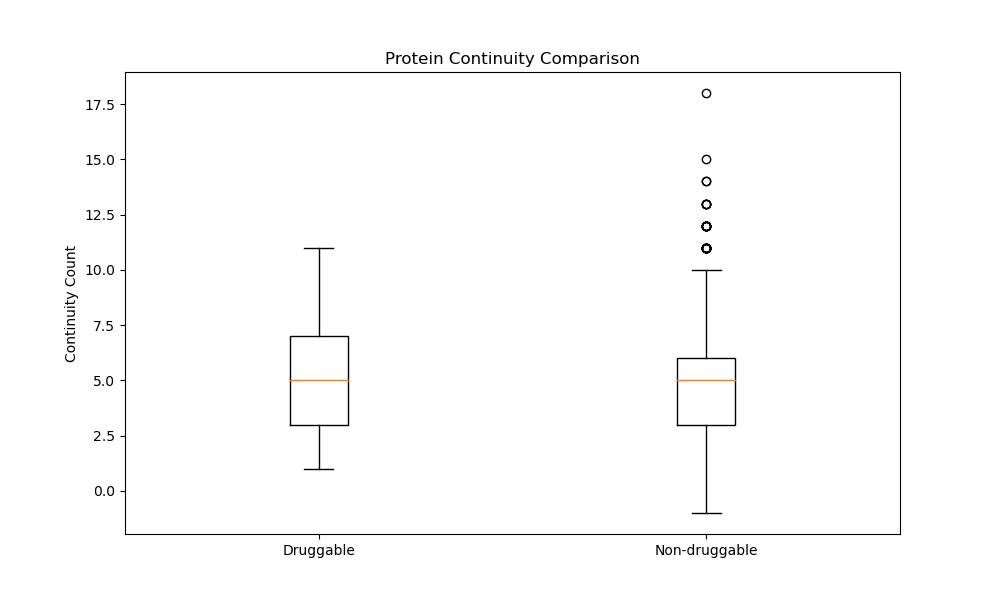


stability
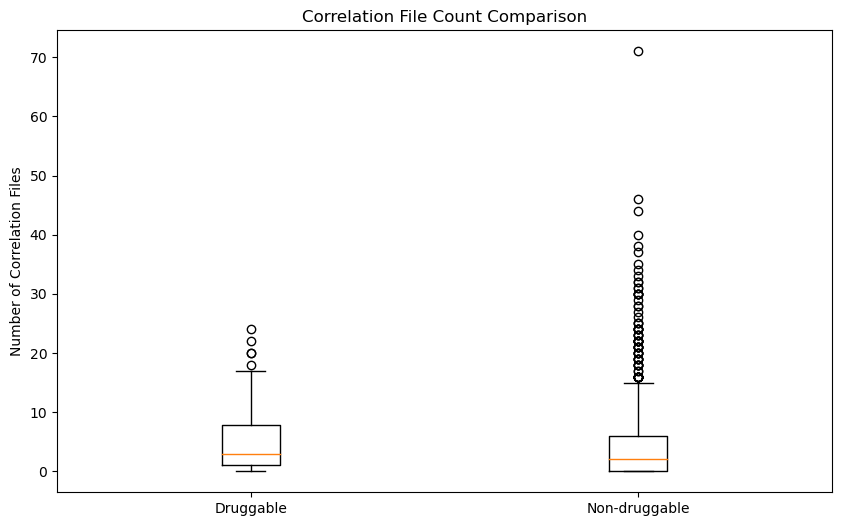
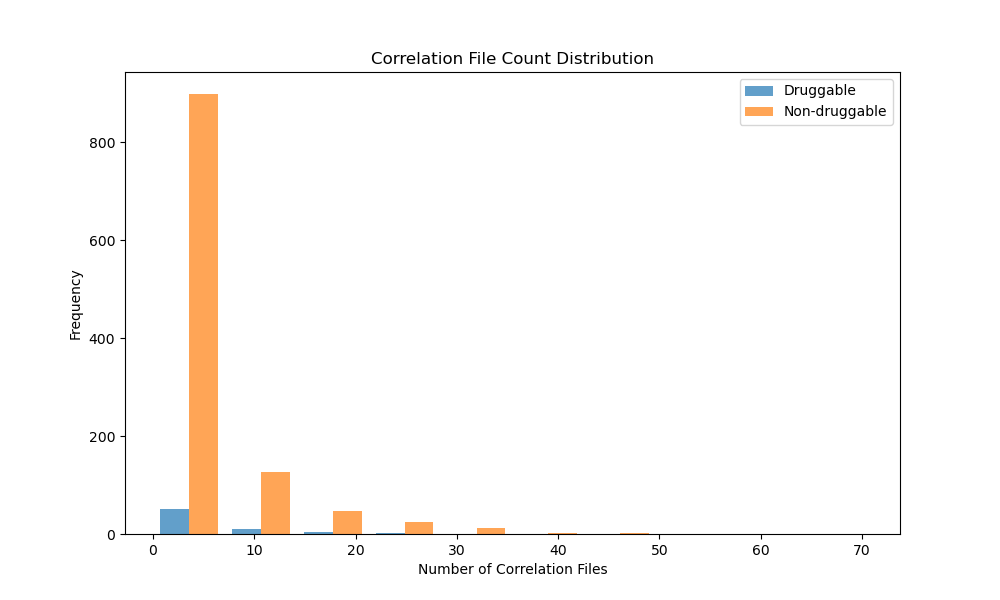
correlation
2025-9-4:cuda_interface的测试文件:test_cuda_interface.py通过。在test路径下,核函数是test_kernel.cu。我非常想吐槽一下,有时候他能跑,有时候跑不了。就cuda时连时不连。测试路径在test_re_4
2025-9-5:开始PocketMatrix测试,测试路径:test_re_5
2025-9-6:在75.1/gxxu/tmp中编译了一下,cuda_ops_test编译成功,说明核函数没有一点毛病,还是参数传递的问题。没有g++他怎么预编译的呢?现在来确定是哪个参数传递的问题。救命啊,我到底干什么,他怎么就跑起来了。但是可以确定的是cuLaunchKernel传入的params一定是指针。
test_re_5/test_pocket_matrix通过,test_pocket通过。///虽然我不知道代码算法是否有变化
开始test_re_6:集成测试,python main.py,和算法修正,新建了输出路径results用于区分output中的参考输出,预计能控制在4min内
2025-9-7:现在是整合一下新的代码。
