2025.10.20-Omnipath(wb脚本)
脚本使用的搜索方式是基于有向调控网络的 “分层上游广度优先搜索(Layered Upstream Breadth-First Search, BFS)”,其核心逻辑与COSMOS “因果路径筛选需按层级控制步数、优先最短路径” 的原则高度一致
Causal integration of multi-omics data with prior knowledge to generate mechanistic hypotheses发表在Molecular Systems Biology
任务:
从下游WB出发,往上搜索三层基因(靶点也许会出现在这些搜索结果中)输出的是根据文献证据和步长排序的基因列表,“把这三层的结果再做D3CARP-AI(TP),得到输出结果进行排序”
先尝试2层,因为layer>3基本会搜索3000以上的基因
/home/data/ydn/2.0/251020_wb/wb/
conda activate pathway2targets_env
Western Blot 差异蛋白:vorinostat.txt
layer:2
min_targets:1
- Rscript wb_upstream.R vorinostat_wb_gene.txt 2 1 vorinostat complete_network.RData
批量运行 bash run_all_wb.sh,样本量大时也可以后台运行
将所有结果提取到/home/data/ydn/2.0/251020_wb/2.0_AI/wb_results/
- Rscript wb_upstream.R vorinostat_wb_gene.txt 3 1 vorinostat
在complete_network.RData/home/data/ydn/2.0/251020_wb/results_all/
| affected_targets | 该上游基因调控的 WB 差异蛋白数(加权后) | 来自 paths_dt 的汇总 | 越多越高 |
| log(total_literature + 1) | 所有路径上文献引用数之和的对数 |
| 文献多 → 高 |
| avg_confidence | 平均置信度分值(very_high=4, high=3, medium=2, low=1) | 1–4 | 置信高 → 高 |
| tf_bonus | 若为转录因子(TF),乘以1.5,否则1.0 | 1或1.5 | TF优先 |
| layer_bonus | 与目标层数成反比,越近越高 |
| 下游层越近越高 |
| net_type_bonus | 网络类型奖励:gene_regulatory=1.3, signaling=1.1, 其他=1.0 | 1.0–1.3 | 调控网络优先 |
| direct_bonus | 若直接调控输入目标基因则×1.3,否则1.0 | 1.0或1.3 | 直接调控优先 |
跑TP-AI(用yj师姐的环境)
75.1
cyj-d3ai
上面环境cyj-d3ai是85.3上面的pytorch_gpu一样的D3CARP-AI的靶标预测按照下面步骤使用,(虚筛需要修改脚本路径)使用:
1、conda activate cyj-d3ai
2、cd path/to/your/file
3、cp /home/dddc/chenyuanjie/hl_wr_tcm_d3carp_calculate/yingshe-d3ai/1-DeepL-TP.sh ./4、bash path/to/your/file/1-DeepL-TP.sh - path/to/your/file/test .smi -m MPNN-CNN
思路:全库跑TP筛选人源与wb_results取交集
上游:
直接跑批量脚本设定
Rscript "$SCRIPT" "$INPUT_DIR/$txt" 2 1 "$base" "$NETWORK"
2 两层 1 上游基因至少要调控多少个目标(过滤噪声用)
AI人源:
Total Mapped Mapped_Percent Human Human_Percent
5901 5900 99.98% 2921 49.50%
交集:AI_TP.sh
输出:/home/data/ydn/2.0/251020_wb/2.0_AI/result/
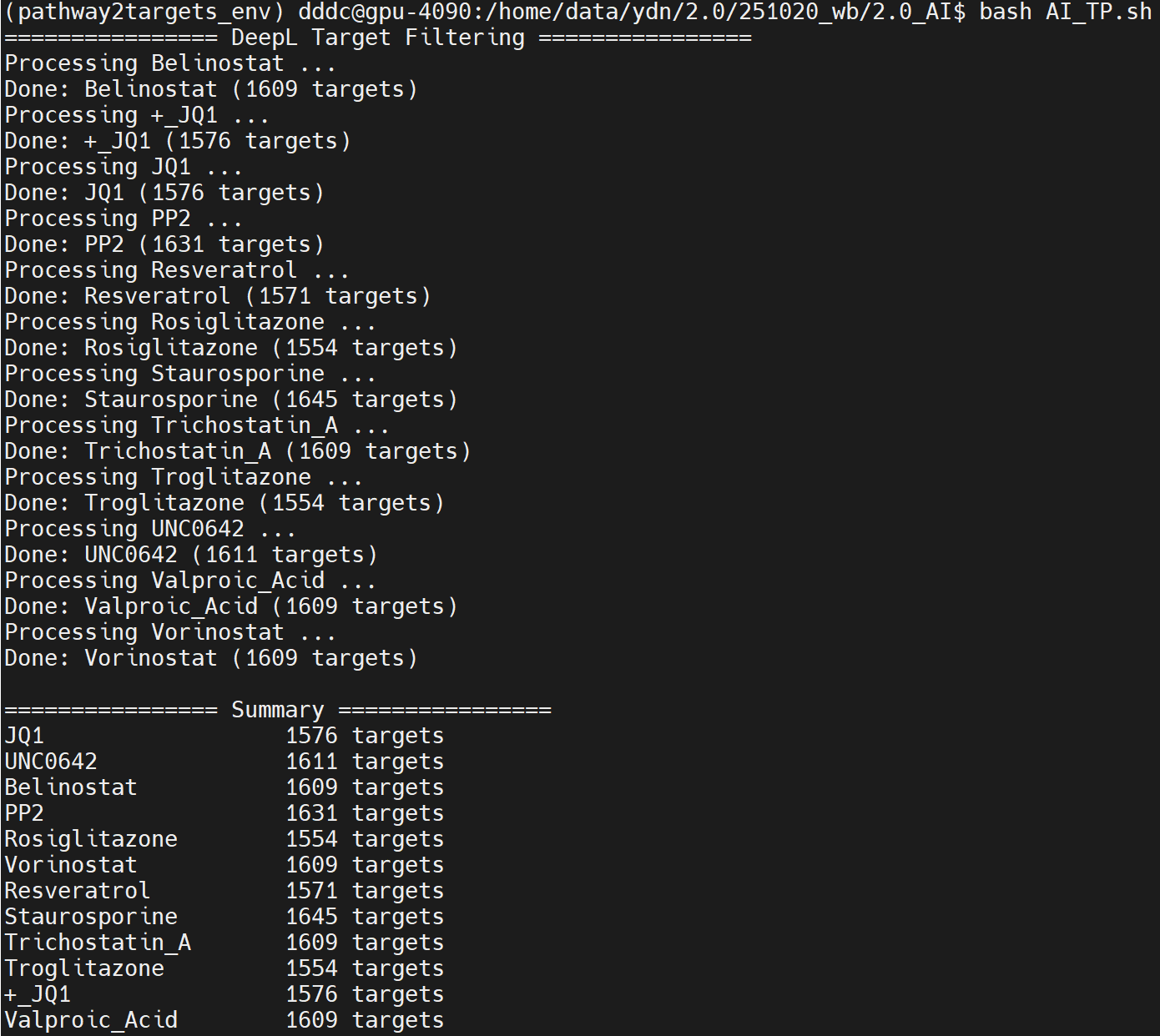
金标准对比:
- Rscript wb_ai_gold_validation.R database.csv result/ AI_result/ "5,10,20,50,100,150"
- database.csv - 金标准数据库文件(需要您提供)
- result/ - WB过滤结果目录
- AI_result/ - AI预测结果目录
- "10,20,50,100" - 可选的Top-K值列表
结果:/home/data/ydn/2.0/251020_wb/2.0_AI/wb_ai_validation_20251028_164408
方法1:Excel 导入法
打开 Excel(不要直接双击 CSV)
点击 Data → Get External Data → From Text/CSV
选择 validation_table.csv
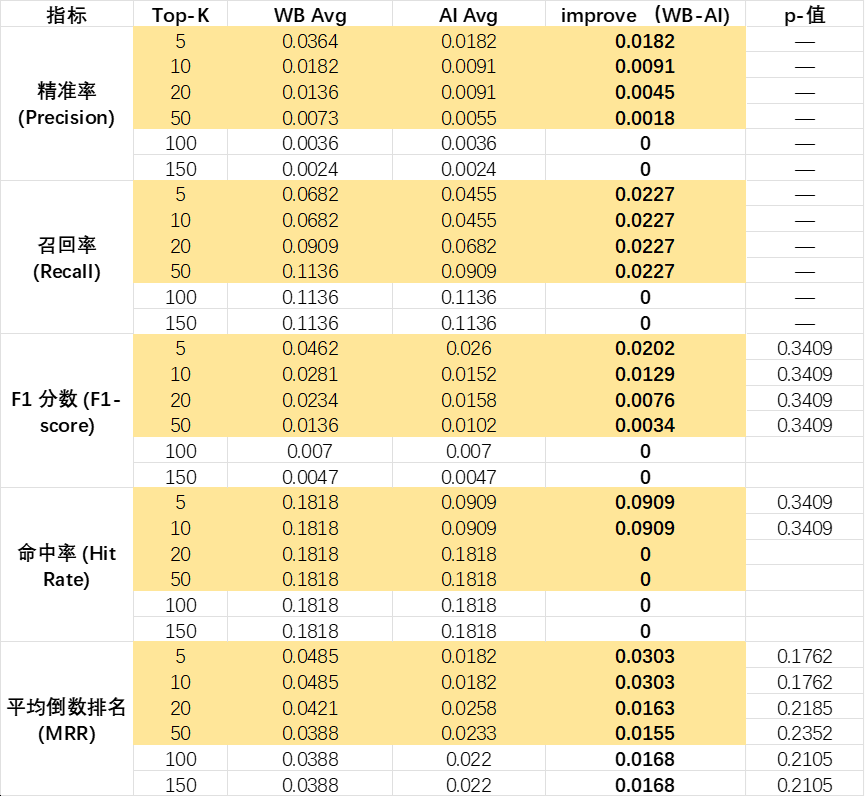
本次基于之前收集的 14 个具有已知靶点信息的化合物,对比了 WB(实验结果)与 AI(深度学习模型预测)的靶点识别性能。综合 Precision、Recall、F1-score、Hit Rate 与 MRR 等指标结果显示:
1、WB 方法在所有 Top-K 范围(特别是Top-5 -Top-50)下的平均 Precision、Recall、F1、Hit Rate 及 MRR 均略高于 AI 模型预测结果。
2、样本数量较少,以及金标准中每个化合物真实靶点数量有限(多为 1–2 个),导致平均值离散分布,显著性较低。
但以JQ1为例AI能找到BRDT,2.0能找到BRD4,取交集以后为空集
有部分GRN基因不在TP库里,怎么做target prediction?
AI-TP人源:1771 GRN:16202 交集:1687
老师建议:1、扩大到3层 2、AI序列最相似相似度统计再做交集,扩大样本。
重新补充人源交叉、3层(还未筛选掉文献少的样本、未考虑相似度)
结果甚至比2层差了,因为没有基因更多了
