Glide共价虚拟筛选
1.前言
2.体系准备
配体部分按《Glide虚拟筛选》反应前的准备
3.共价对接
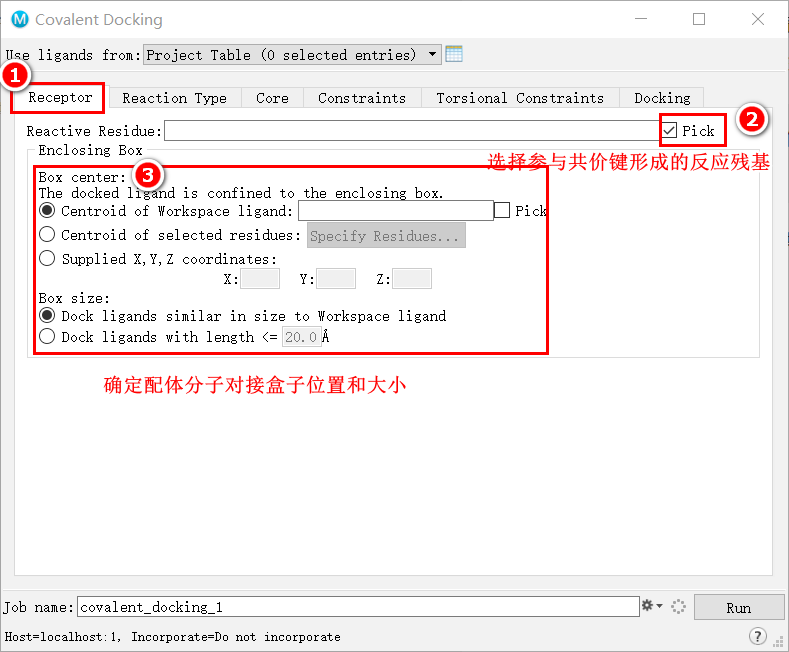
Covalent Docking
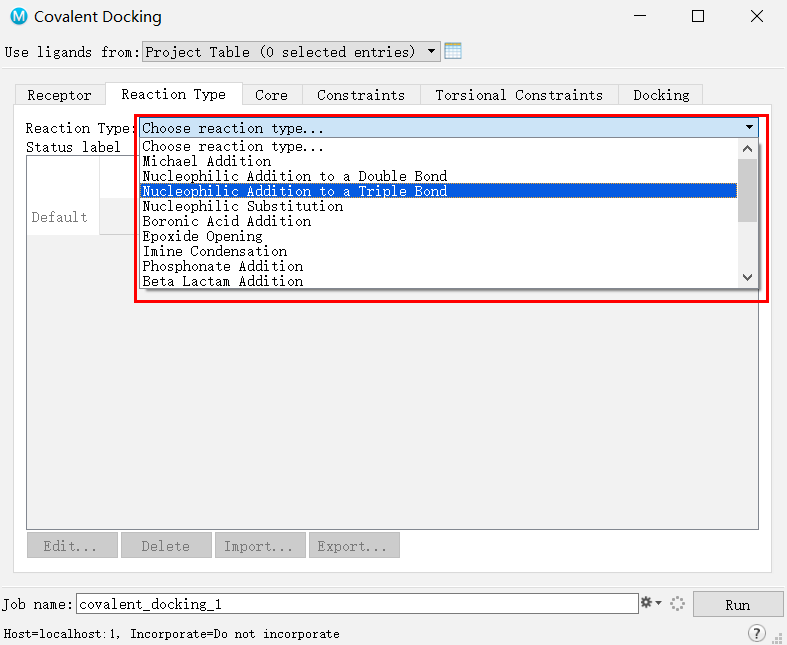
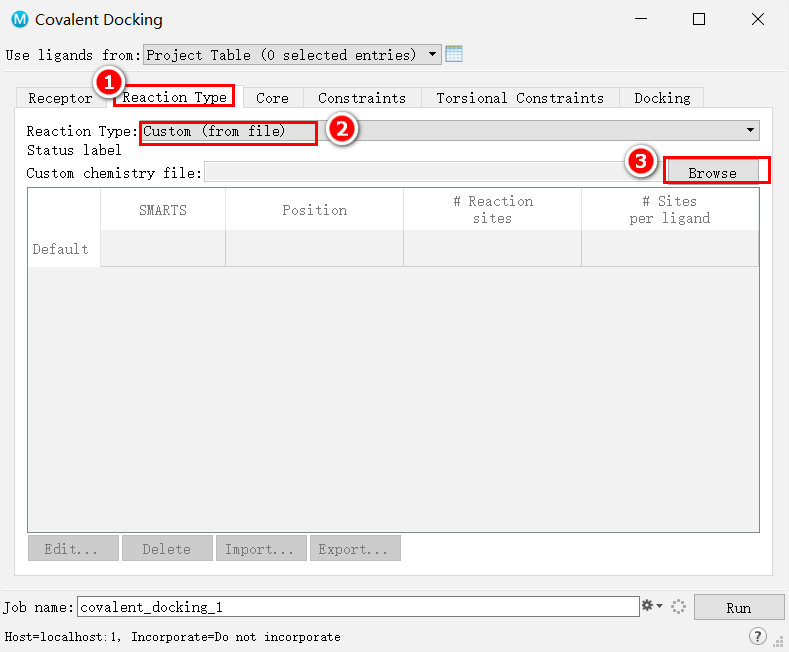
如果没有找到需要的反应类型,可以选择从官网下载其他反应类型,并从外部读入:
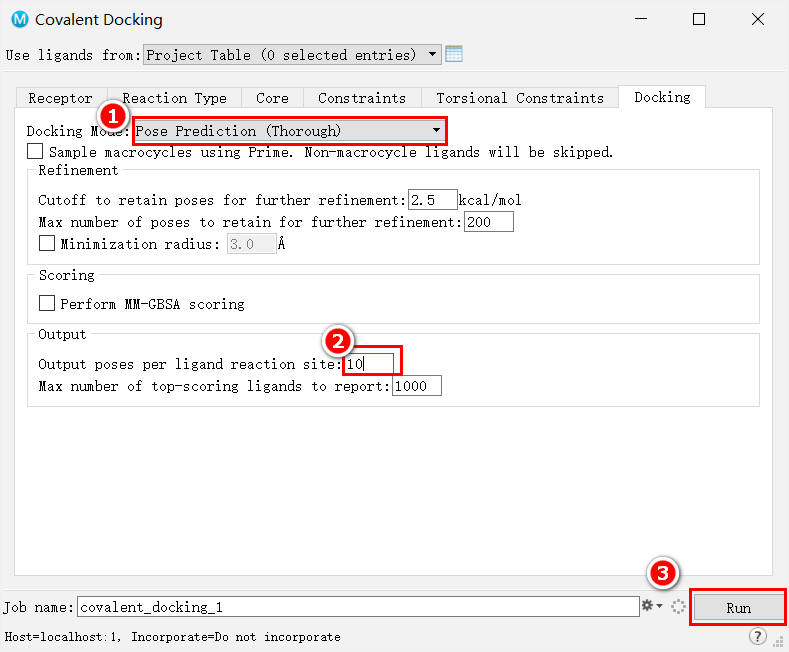
注意,这里设置好以后要在左侧列表中多选选中对接体系和准备好的分子,再run
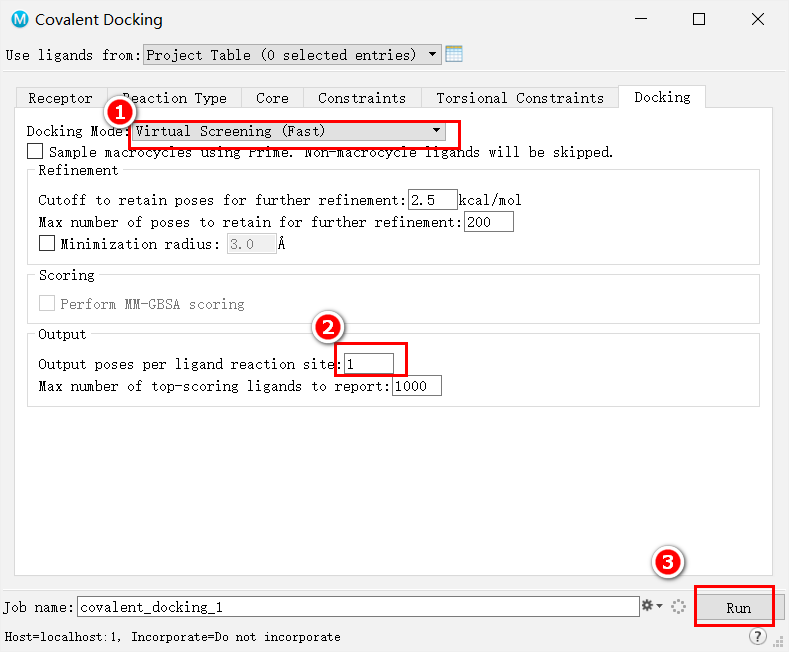
实际使用时Fast一直报错,Thorough跑通。
4.数据处理
阅读下面的数据处理方法时,作者默认读者已经熟悉《Glide虚拟筛选》一文的各种处理方法。
使用服务器的maestro打开对接结果文件,选择docking score排在-8之上的(阈值自主选择),sort 内选择按MOLNAME分组(实际上就是按分子名分组),选择每组第一个,select,invert select反选,删除其他的,导出为first.maegz文件。在property tree中去掉多余的内容,最好只保留MOLNAME,CAS,docking score列(否则pymol打开会很卡),导出为first.txt文件,再导出一份maegz文件,作为pymol后续看结构的pse文件。
接下来使用脚本从库中提取对应sdf文件:
使用方法:
- python glide_covalent_docking_mergy.py -s DRN.sdf -g first.txt -r NucleophlicSubsitution -o result.sdf
-s 对接前结构的SDF文件,其MOLNAME一项为分子名。
-g maestro产生的txt文件
-r 输入你的反应类型
-o 输出文件名称
maestro载入sdf文件,聚类,选择所需的数目的分子,删除其他分子,导出为xlsx即可。同时first.maegz文件手动保留对应的复合物记录,将MOLNAME与docking score合并写入title,在property tree中去掉多余的内容,最好只保留MOLNAME,CAS,docking score列,导出properties项选择shown,否则pymol会卡死。
