pymol一些操作记录
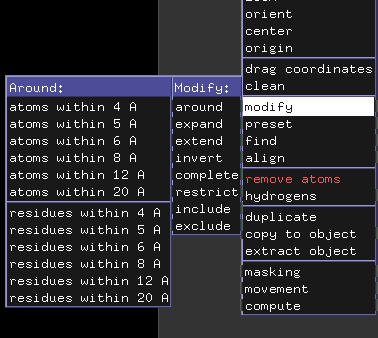
选择5A 范围内的氨基酸
图形界面选中配体,A-modify-around 5A residues
select near_residues, byres (9ayk within 5 of obj01)
这是小分子为obj01,蛋白为9ayl(里面小分子已扣掉)
pymol中文教程:
pymol license:
选择一条链,除去其他的
- select chain_A, your_protein and chain A
- create new_protein, chain_A and polymer
创建一个项目,包含某一段(先选上,再创及命名)
select new_obj, protein and resi 5-45
create new_protein, new_obj
成组:
group my_group, obj1 obj2 obj3
group num108_10.20,num1 rep.c108 complex108 complex108_pol_conts obj06 measure12 measure13 measure14
解散组:用ungroup my_group,然后变成对象加组的名字,再删除组。如果是delete my_group的话,会直接删除组以及组内对象。
往已存在的组内添加对象:
group mygroup, add=measure05 measure06 measure07 (不能使用measure05-07)
- reset
在 PyMOL 的命令行中输入 reset,这会将当前视图重置为默认视图,通常情况下,蛋白质会回到可见区域
- dss
用于在蛋白质结构中计算并显示二级结构
去水、去甘油、加氢,去d3pocket找口袋
处理蛋白:
- create 4ztbz_pr,4ztb and polymer
再add hydrongens
选择残基,加标签,显示为sticks
- select resi 478+548
select activate_site, resi 478+548
select activate_site-ca, activate_site and name CA
label activate_site-ca, resn + resi
show sticks, activate_site

选择某一个保存
set state,18
save testpymol.pdb,0.12_skip20clusters, state=18
- disable all
- split_states clusters
- delete clusters
- /for i in range(3,100): cmd.delete( "clusters_%04d" %i )
- dss show cartoon
- util.cbam clusters_0002
- align clusters_0001 and ss h, clusters_0002 and ss h
单双键

绘图
pengziyu-teakki:
主要是下面这个图用来保存做最终图所需要的图,教程前面的步骤可以自己安排

由于参数的顺序与 Select 命令(其中所选内容的所需名称是第一个参数)不同,因此将其视为类似于“mv”shell 命令可能会有所帮助,其中首先列出现有对象,其次列出目标。
set_name sele,lig
select res, resi 8+9+6+39+77+78+74+41+44+79
hide
show sticks,lig|res
hide sticks,h.
zoom lig|res
color wheat,lig & name C*
set label_size,20
pi-pi stacking堆积
选择viewing模式,select为atom模式
运行脚本center_of_mass
com sele,object=p1
set sphere_scale,0.25
(把球变小)
测量wizard--measurement
氢键
create complex, 20085 rep.c3
A--preset--ligand sites--cartoon,即显示出键,然后再测量wizard--measurement
显示出measure01.。。
sele resi 8+78+4+5+lig
A--find--polar contacts--excluding solvent
Transparency透明度
- set transparency, 0.65, i. 1-100
操作:
setting--transparency--cartoon--0.5
重命名:
cmd.set_name("measure02", "pi-pi_stacking02")
或
set_name 旧,新
B因子计算:通过分析电子密度图和模型之间的匹配程度,计算每个原子的B因子。B因子反映了电子密度的模糊程度,从而反映了原子位置的确定性或不确定性。
重新编号:
注意编号问题,有时候操作是从1开始的,最后返回到原始编号
help alter
alter all, resi=str(int(resi)+100)
对所有重新编号,在原有基础上加100

rebuild
select res,resi 8+78
color blue, name C+N+CA
color red, sele 再sticks
new

隐藏氢原子
- hide (hydro)
这个命令会隐藏当前所有显示的氢原子。 hydro 是 hydrogen 的简写,指代氢原子。如果有其他显示的内容(例如原子、分子等),这些内容仍会显示出来,只有氢原子会被隐藏。
调节小分子变换速度

