一些教程或工具
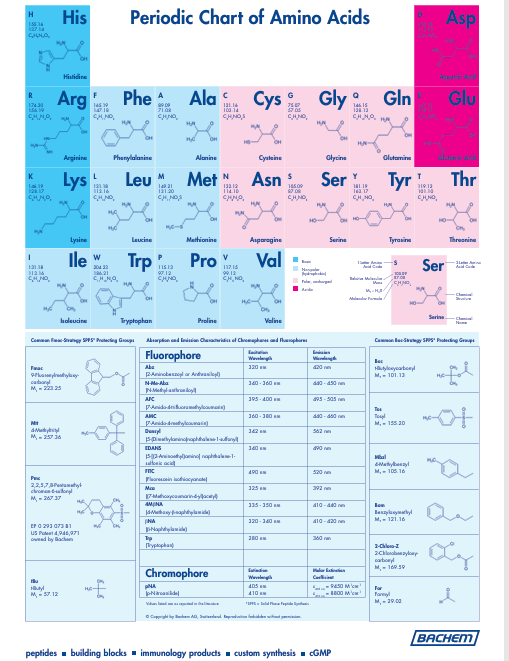
蛋白质表-还比较清楚的:
对接:
smina对接
(见对接教程)
glide-sp/xp等
(见对接教程)
共价对接:
RDKIT
(还没试)
smiles-分子语言
序列比对和rmsd
当两条蛋白质序列的相似度大于30%时,他们的三级结构以及生物学功能也可推测为很接近的。
序列比对(蛋白序列blast):
D3POCKET 服务器使用:
23服务器可以
D3CARP(teakki上教程)
服务器上操作:
lrt:
另外75.3上也有similarity
USAGE: sh ${absolute_path}/1-Ligand_Similarity_2D_TP.sh -l [ligand](sdf, mol2, mol or smi) -f [fingerprint](FP2, FP4, or MACCS) -d [cutoff] -c [cpu]
激活环境-rdkit
cp /home/dddc/ydn/d3carp-similarty/1-Ligand_Similarity_2D_TP.sh ./
75.1上面也有similarity,环境rdkit_y,用法一样
hzj:
d3ai-TP-其他服务器上
药物靶点:
小分子靶点预测
GROMACS-常规MD
zotero教程:
decoys
DUD-E
对小分子生成decoys(50个)可用的网站(生成的是smiles格式,发送到你的邮箱一个压缩包),有时候可能会不发送,过段时间就好了:
还有其他方法待补充……
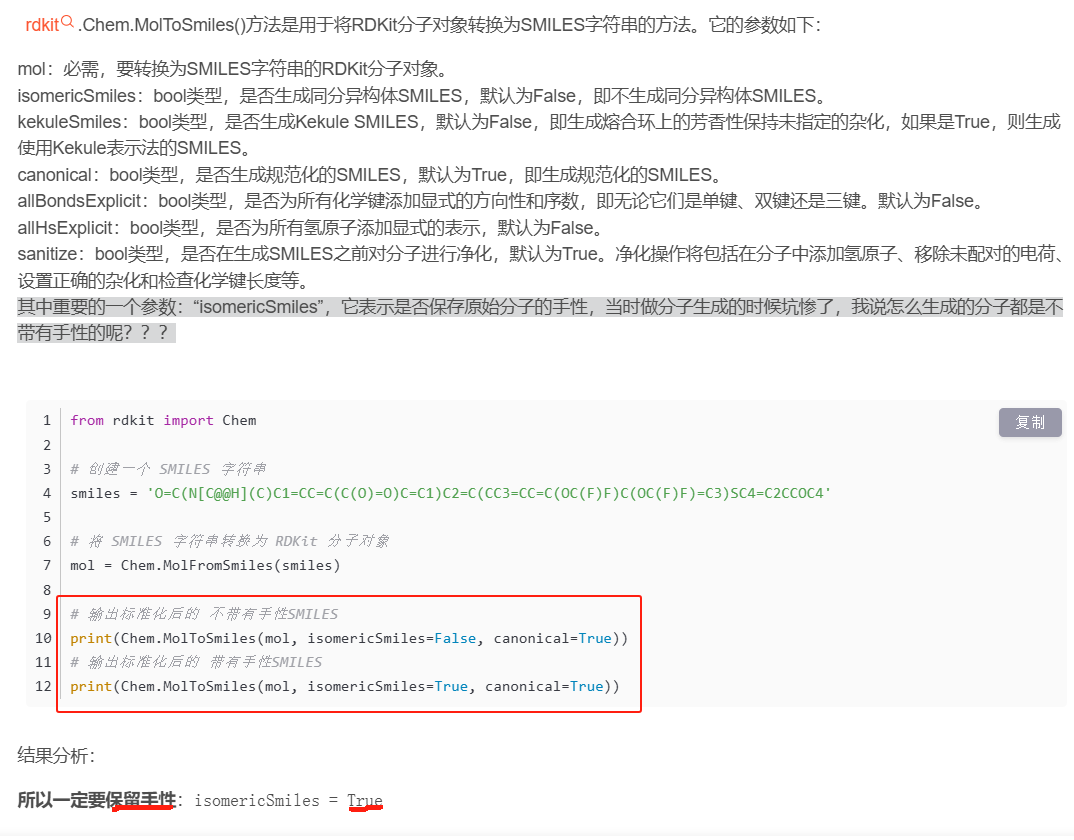
蛋白-配体2D相互作用工具汇总
protein plus(网页版):可以看二维相互作用ligplot
ligplot(需下载软件):
应该是每年会过期一次需要重新搞licence或者下载
分子对接 二维可视化软件 ligplot 的下载安装与使用 链接:https://pan.baidu.com/s/1483MhfehugI8u0lNqRtu1Q_哔哩哔哩_bilibili
修改好路径为下面的可链接到pymol

pymol+ppt作图教程:
ppt保存高分辨率图片
方法1:
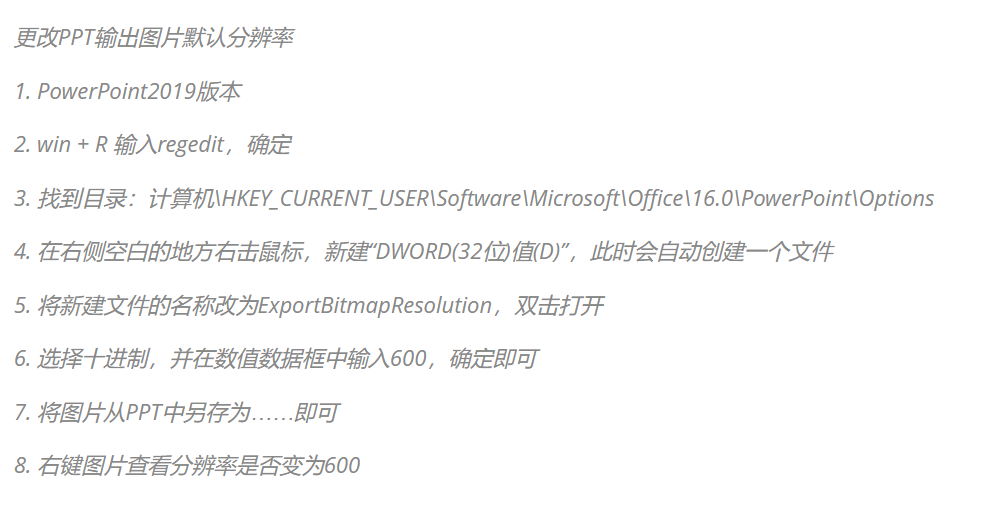
方法2:
插件iside
安装后可在ppt直接保存导出多张图片
pymol观察氢键:
选中小分子配体后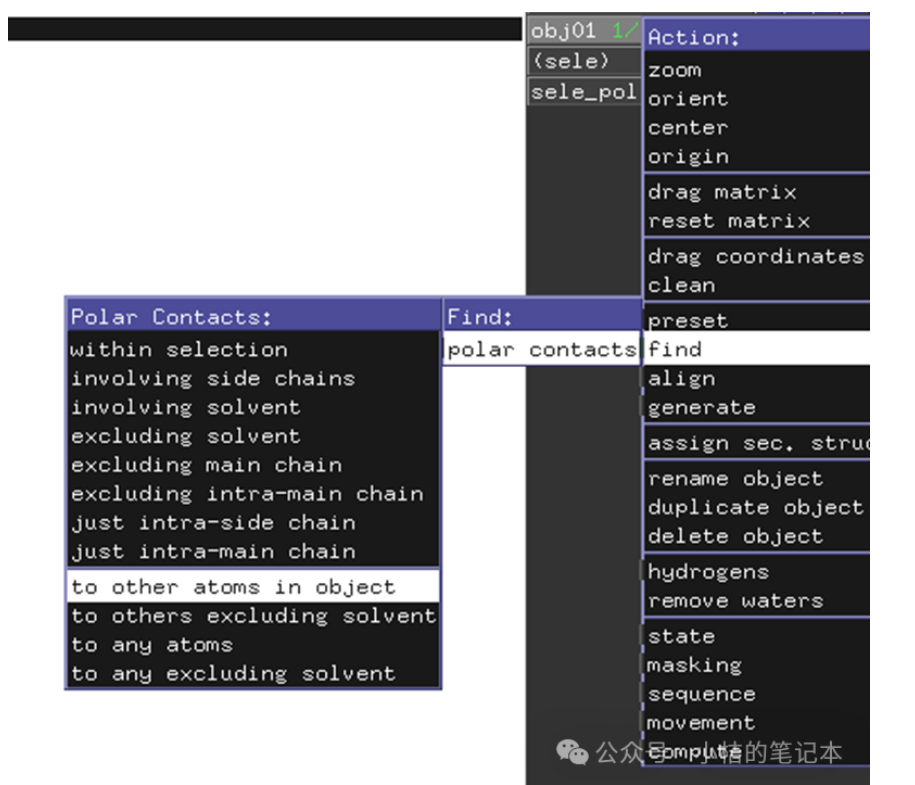

或者
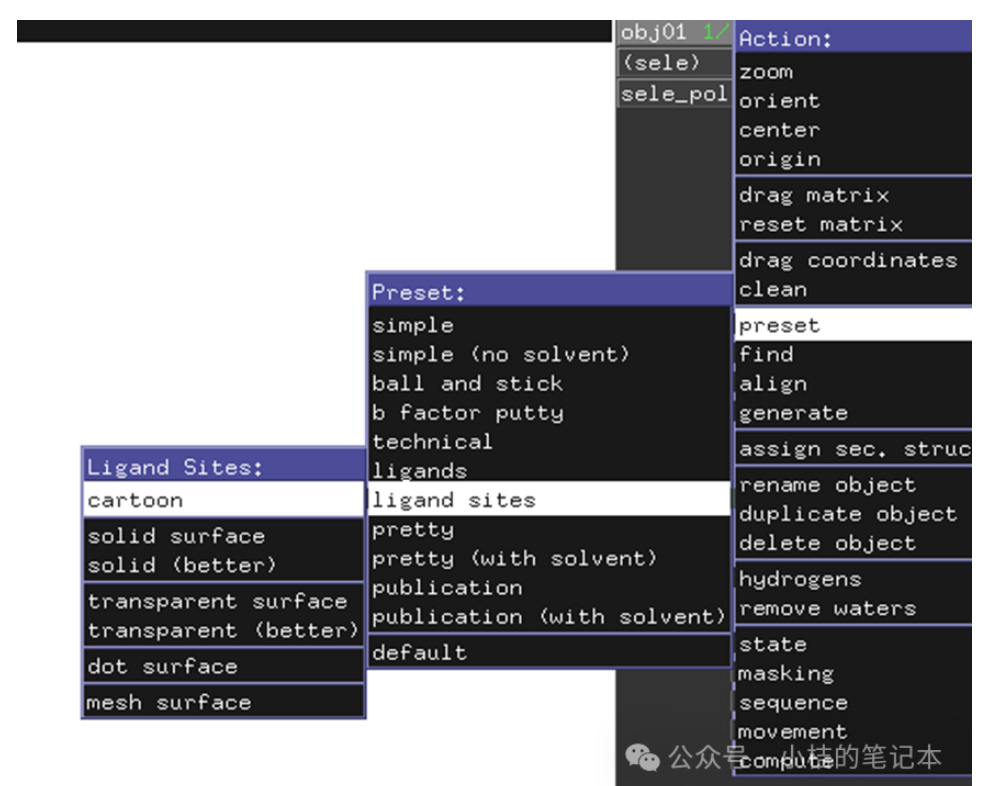
氨基酸突变
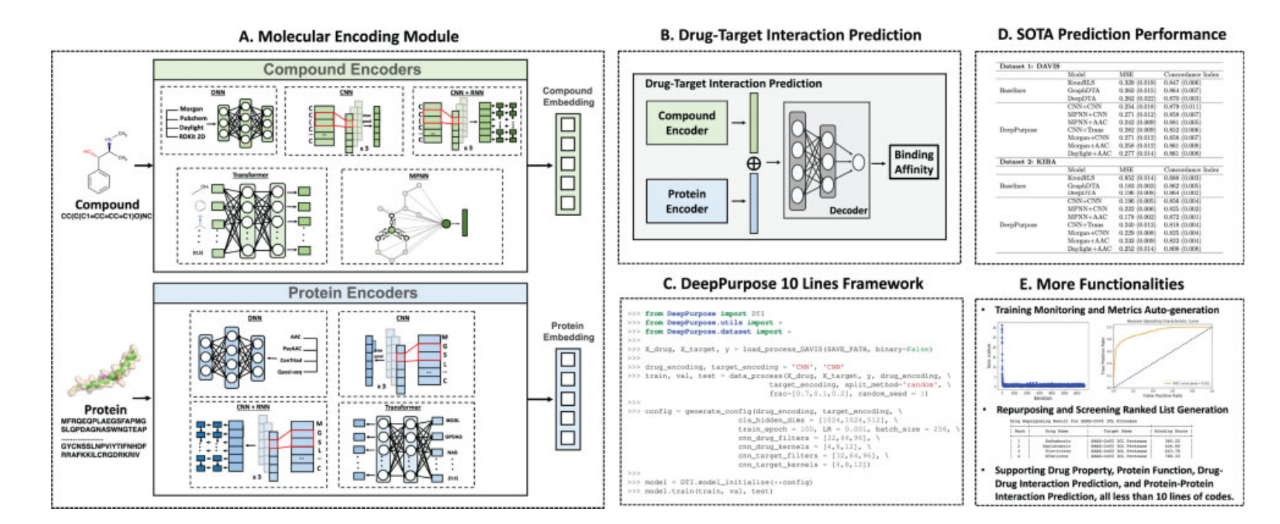
deepurpose
python画图
待看
plt.savefig('my_plot.png')
安装vscode
挺详细的
中断脚本:
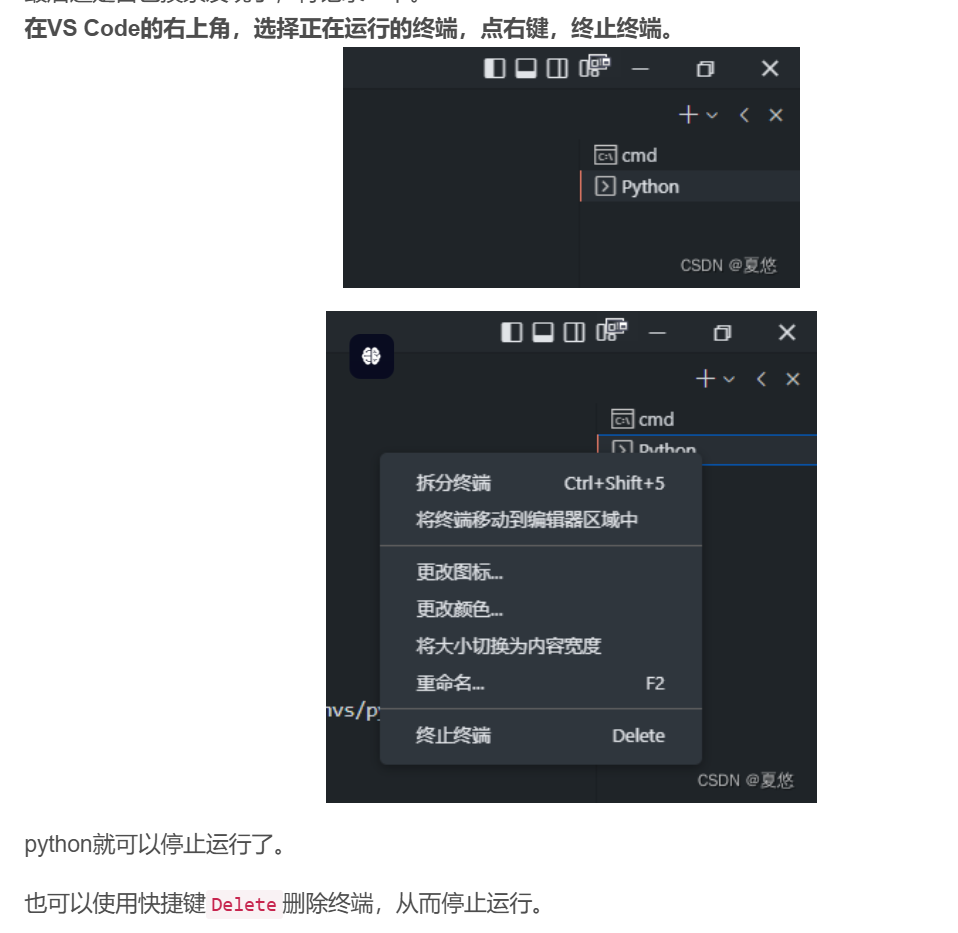
调出终端:

集合相似性:

配置pytorch_gpu
师姐:
在75.1 4090上装一直报错可能是conda问题,之前安装没有问题
github
必须和git建立连接吗?可能是有两种上传方法:直接通过官网和借助git
endnote下载安装
小红书教程:(国科大版-需要中国科技云账号)
55 🍊橘狸(生信、数据分析学习中)发布了一篇小红书笔记,快来看吧! 😆 VEcWddZycOwdA9Q 😆 http://xhslink.com/m/3C7Va883kgi 复制本条信息,打开【小红书】App查看精彩内容!
新机房打印机
(docx教程是台式有线的)
IP:172.21.150.201
掩码:255.255.255.0
网关:172.21.150.254
