Pymol常用命令
选择命令
select resn HIS 按照残基名称选择
alter (chain D), resv += 223 修改残基编号,依次顺移223位
help + 命令 帮助文档
alter name, chain ="A" 加链的信息
select all
create sele, com 合成一个PDB
select idx. +原子序数
select name +原子名称
寻找蛋白1qox互作界面(chainI和J)的残基
- interfaceResidues("1qox", cA="c. I", cB="c. J", cutoff=0.75, selName="foundIn1QOX")
脚本批量叠合算重原子的RMSD
- from pymol import cmd
- import pandas as pd
- rmsd_df = pd.DataFrame(columns=["name","rmsd"],index=[i for i in range(1,76)])
- cmd.load("ranked_1_0.pdb")
- cmd.create('ali1','resi 1-125 and ranked_1_0')
- inde = 1
- for i in range(1,16):
- for j in range(5):
- name = "ranked_" + str(i) + "_" + str(j) + ".pdb"
- cmd.load(name)
- selection="resi 1-125 and " + "ranked_" + str(i) + "_" + str(j)
- cmd.create('ali2',selection)
- a = cmd.align('ali1 and name CA+C+N+O','ali2 and name CA+C+N+O')[0]
- rmsd_df.loc[inde,"rmsd"] = str(a)[:5]
- rmsd_df.loc[inde,"name"] = "ranked_" + str(i) + "_" + str(j)
- inde += 1
- cmd.delete('ali2')
- rmsd_df.to_csv("rmsd.csv",index=False,encoding='utf-8')
找出蛋白的表面氨基酸:
在质心放虚原子
#将center_of_mass.py放在pymol安装目录下的lib文件夹
import center_of_mass
#选中要创建质心的原子
com sele,state=r1
#修改质心球大小
set sphere_scale,0.4
指定RGB颜色着色
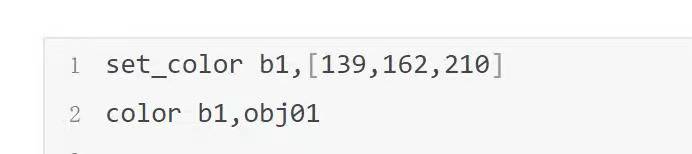
生成静电势表面
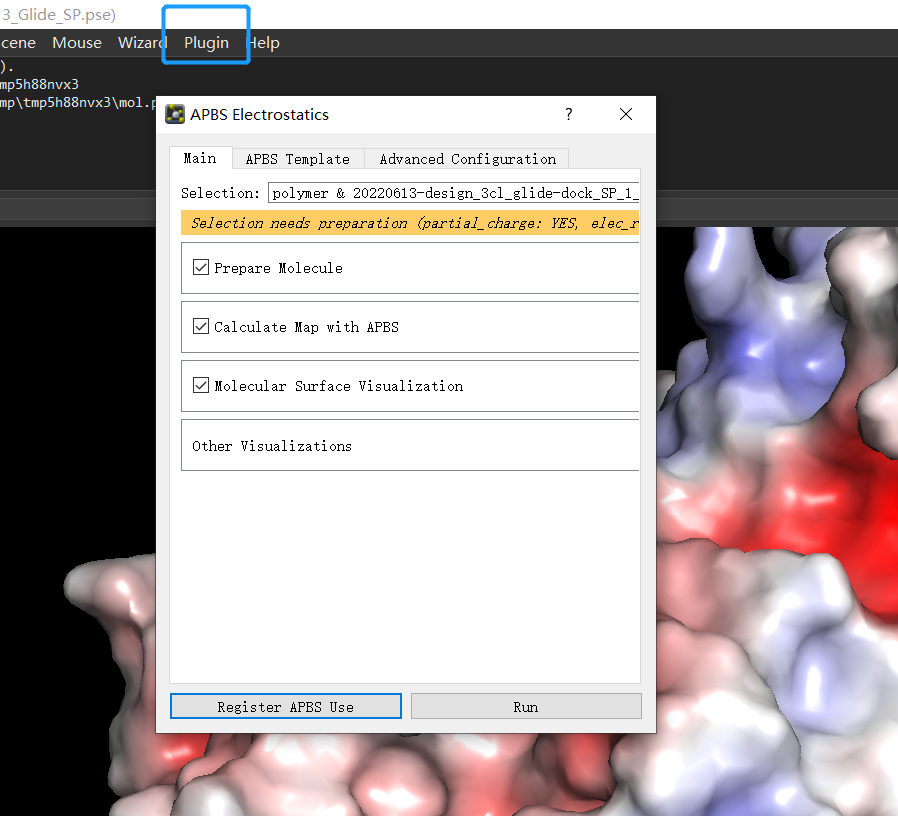
开源版本中,APBS 使用单机版,pdb2pqr 使用在线版(需提前计算并下载 pqr 文件,当然也可以使用单机版 pdb2pqr),剩余步骤按照 APBS Tools2.1 插件说明即可。
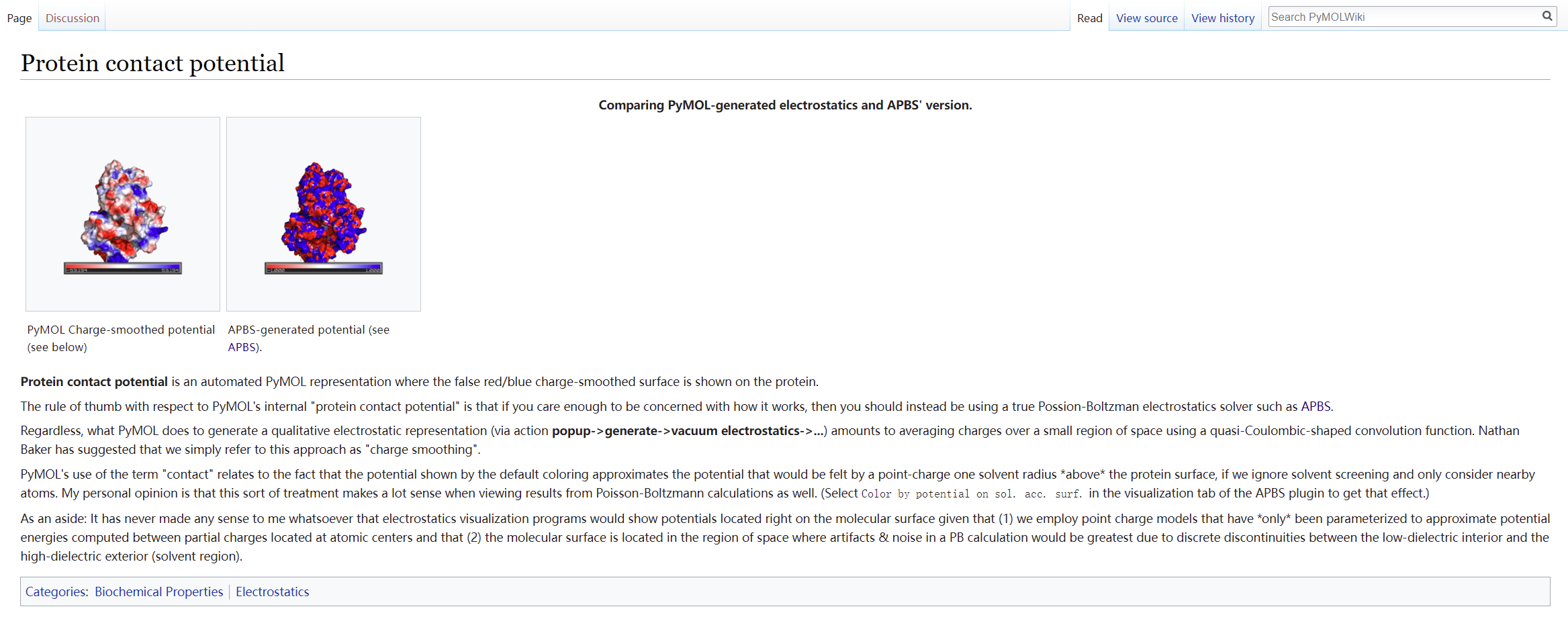
在 PyMol 中,还有一种方式为 Action -> generate -> vaccum electrostatics 可以计算 “smoothed charge”,与静电势定性一致。详细描述可参考 Protein contact potential。
选择语法

pymol选择的时候,默认主链上的N都是氢键供体,主链上的O都是氢键受体
