0702 D3Docking
结果
数据量还是很大的
库中的蛋白都和99个老药进行过对接,那自然就有99个打分,我们新对接的化合物的打分纳入这99个,排个序
- # 打分排名第一名的挑出来
- for a in
`cat protein.txt` - do
- for i in
`cat index` - do
- #awk '$9<6{print}' ../results/${i}/${a}/${a}-${i}.txt|sed "s/^/${a}; ${i}; /g" >>result.txt
- awk '$9<2{print}' ../results/${i}/${a}/${a}-${i}.txt|sed "s/^/${a}; ${i}; /g" >>result.txt
- done
- done
- # 这其中,28个分子在至少一个靶点的某一个口袋打分能排到第一
- (base) [zjxu@R820 analysis]$ awk -F ';' '{print $2}' result.txt |sort|uniq|wc -l
- 28
- # 仅从打分来看,信息比较集中
- # 先排序(打分在第5列)
- # 很集中,前10个打分结果 实际都是两个化合物在不同靶标/口袋上的打分
- (base) [zjxu@R820 analysis]$ sort -t ";" -k 5,5n result.txt |head|awk -F ';' '{print $2}' |sort|uniq
- TCMID-19532
- TCMID-28188
- # 让我瞅瞅他们的结构,相似性以及D3AI结果
- # 结构
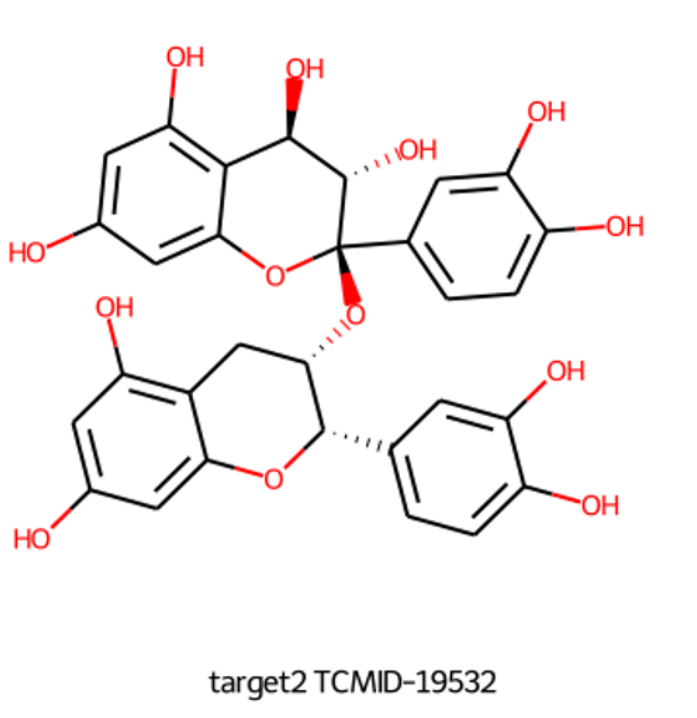
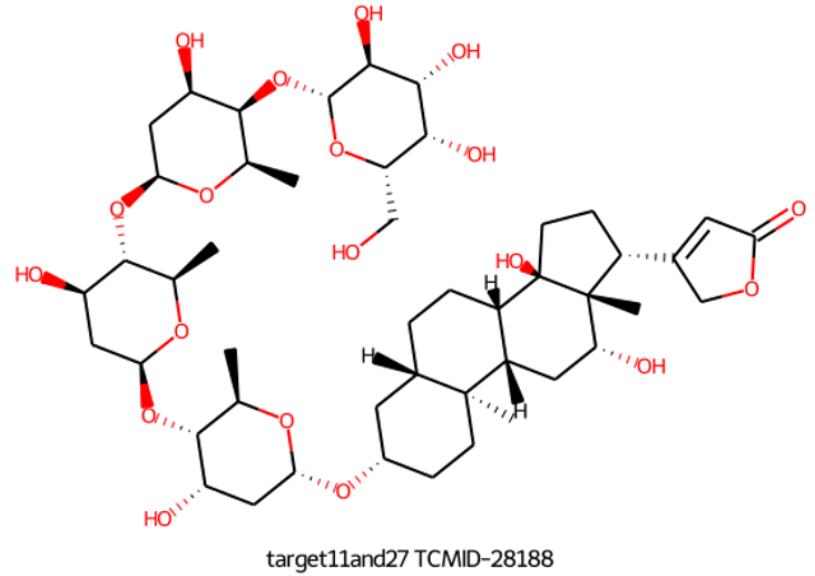
- 感觉羟基过多,打分会被高估,
- 下图左边这个分子排在所有分子的第5位;右图这个第
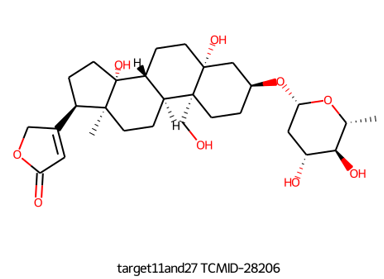
- # 相似性结果
- grep TCMID-28188 d3S-result.txt
- 部分化合物 如 TCMID-19532 对应的阳性化合物 ICV737 D3Target 库里没有(网站上没有记录)
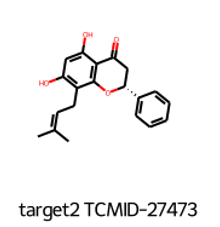
- TCMID-27473 不错, 结构较简单, 且和目前报道的阳性化合物结构并非完全一致
- target2 TCMID-27473 ICV283 0.913043 0.64787
- target2 TCMID-27473 ICV713 0.962963 0.60831
- # AI 结果
- # 172.21.85.3 /home/yqyang/zzy/hsbd/d3ai-cov
- grep TCMID-27473 workdir/*/* >TCMID-27473
- awk -F '|' '{print $2,$4,$5}' TCMID-27473|sort -k 3
- 并不好,但是可见,再SARS_Pap上的结果还说得过去
- # 再看回打分 172.21.85.12 /home/zjxu/zzy/hsbd/dock/d3S-36mol/0702-2pro/analysis
- (base) [zjxu@R820 analysis]$ sort -t ";" -k 5,5n result.txt|grep -n TCMID-27473
- 69:ORF1ab_819-2763_Papain-like_proteinase+Dimer; TCMID-27473; Pocket2; score; -10.53
- 71:ORF1ab_819-2763_Papain-like_proteinase+Dimer; TCMID-27473; Pocket3; score; -10.52
- # 我删了后面三个数据(原子效率,原子量效率,排名(都是1))
- # 对比下最高打分,差的不算远,-10 也够高了
- (base) [zjxu@R820 analysis]$ sort -t ";" -k 5,5n result.txt|head
- ORF1ab_3264-3569_3C-like_proteinase+Dimer; TCMID-19532; Pocket1; score; -14.00
- ORF1ab_3264-3569_3C-like_proteinase+Dimer; TCMID-19532; Pocket2; score; -13.97
- # 看结合模式
- cp /home/zjxu/xbzhang/2019-nCov-final/preparation/protein-v1/ORF1ab_819-2763_Papain-like_proteinase+Dimer.pdbqt ./
- cp ../../dock-conf/TCMID-27473/ORF1ab_819-2763_Papain-like_proteinase+Dimer-TCMID-27473-pkt2-0.sdf ./
- cp ../../dock-conf/TCMID-27473/ORF1ab_819-2763_Papain-like_proteinase+Dimer-TCMID-27473-pkt3-0.sdf ./
- 大致看了下,相当好的结合,回家仔细把相互作用分析下
- 还有个问题是pkt2,pkt3实际上对接到了相同的位置,检查后发现 pkt3 包在 pkt2 外面
- 对我这个影响不大,如果跑MD,大概率是用 这个 体系了‘

- 还有个小问题,这玩意的受体,是swiss-model建出来的。。。,不是晶体结构
- 写文章,是可以说用的是D3Docking 库中的结构,其本质是基于SARS-CoV的单体结构(PDB ID: 5Y3E) 同源建模得到的二聚体
记录
- /home/zjxu/zzy/hsbd/dock/d3S-36mol/0702-2pro
- # 把D3Similarity得到的36个分子重新命名后的一个个mol2文件cp过来
- for i in
`cat 36index`;do c=`grep $i pro-mol.txt`;sed -i "2s/.*/$c/" 36/$i.mol2;done - # pre-dock.sh 的脚本改一下即可(路径以及要支持mol2输入)
- # 其余脚本照搬即可,index(化合物)和protein.txt是重点
- # D3Similarity预测中有5个靶标,实际只能找到2个
- (3C-like protease 和 Papain-like protease)
- # 这两个靶标对应12个蛋白构象,90个口袋,估计明天早上能有个初步结果
- # 其余靶标还得找
- (base) [zjxu@R820 0702-2pro]$ nohup sh do.sh &
- [1] 28142
- 一个晚上完成了(少于12小时)

