12.4 网药
成分信息收集
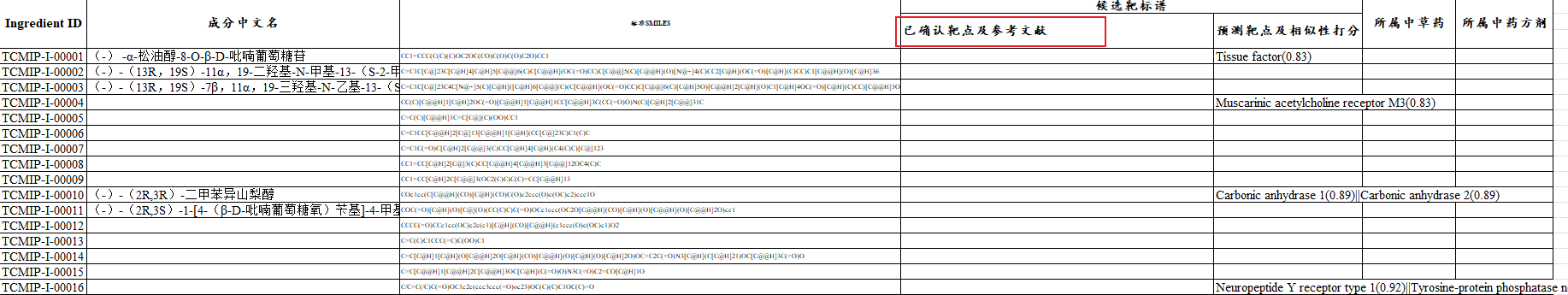
D3AI预测有902个化合物是化湿败毒方中发挥潜在活性的靶点
靶点信息收集
- # 将这902个化合物smiles拿出来,到ETCM数据库中寻找他们的已知靶标
- # 85.3 /home/yqyang/zzy/hsbd/np/get-hsbd-mols-targets/d3ai-902
- # etcm-mols-smi-targets-fyl.csv 源自许海玉老师组的付宇蕾师姐提供的数据,包含了确认的靶点信息
- # 阅读文献可知,确认的靶点信息源自BindDB 数据库
- # 根据902个化合物的smiles式,提取对应的行,找出确认靶点信息
- for i in
`cat d3ai902mol-smi.txt`; do grep -w "$i" ../../data/etcm-mols-smi-targets-fyl.csv | awk -F ',' '$3!=""{print $3}' >> d3ai-902mol-targets-sure.txt;done - # csv文件中,第三列若记载了多个靶点,彼此间用“||”隔开,故文本操作将其替换为换行符
- # vi :1,$s/||/\r/g 将“||”替换为 换行符
- # 去重后,共有156个靶点
- (base) [yqyang@localhost d3ai-902]$ awk -F '(' '{print $1}' d3ai-902mol-targets-sure.txt|sort|uniq > raw-d3ai902mol-156targets
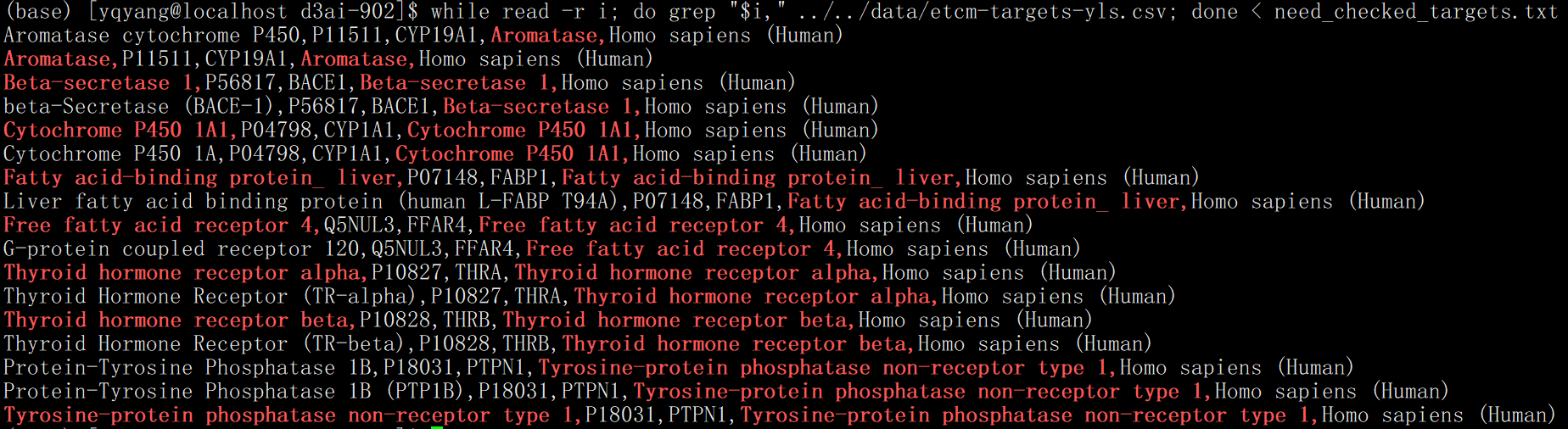
- # 预实验:根据靶点名称提取UniProt ID,通常情况下,每一个名字应该只对应1个id,检查不是1的结果
- # 遍历输入文件
- while read -r i; do
- # 使用 grep 查找匹配行,并计算匹配的行数
- line_count=$(grep -c "$i," ../../data/etcm-targets-yls.csv)
- # 如果行数不为1,将 $i 输出到 unmatched_file
- if [ "$line_count" -ne 1 ]; then
- echo "$i" >> need_checked_targets.txt
- fi
- done < file/raw-d3ai902mol-156targets
- while read -r i; do grep -c "$i," ../../data/etcm-targets-yls.csv; done < need_checked_targets.txt
- # 手动修改 raw-d3ai902mol-156targets
- # 删除的靶点名称:D(明显没这个靶点)
- # 其余靶点只是重名,id是一样的,无妨
- mv file/raw-d3ai902mol-156targets file/hsbd-d3ai-check-155targets
- # 要统计对应的
- while read -r i; do grep "$i," ../../data/etcm-targets-yls.csv|awk -F ',' '{print $2}'>> hsbd-d3ai-ETCM-targets.txt; done < file/hsbd-d3ai-check-155targets
- # 从Genecards上下载“SARS-CoV-2”相关蛋白(excel),共7096个有UniProt ID
- # 与HSBD-D3AI 成分潜在靶点取交集
- cat genecards-SARS-CoV-2.txt hsbd-d3ai-ETCM-targets.txt |sort|uniq -d >insert
- (base) [yqyang@localhost d3ai-902]$ wc -l *
- 902 d3ai902mol-smi.txt
- 0 file
- 7096 genecards-SARS-CoV-2.txt
- 155 hsbd-d3ai-ETCM-targets.txt
- 75 insert
- 8228 总用量
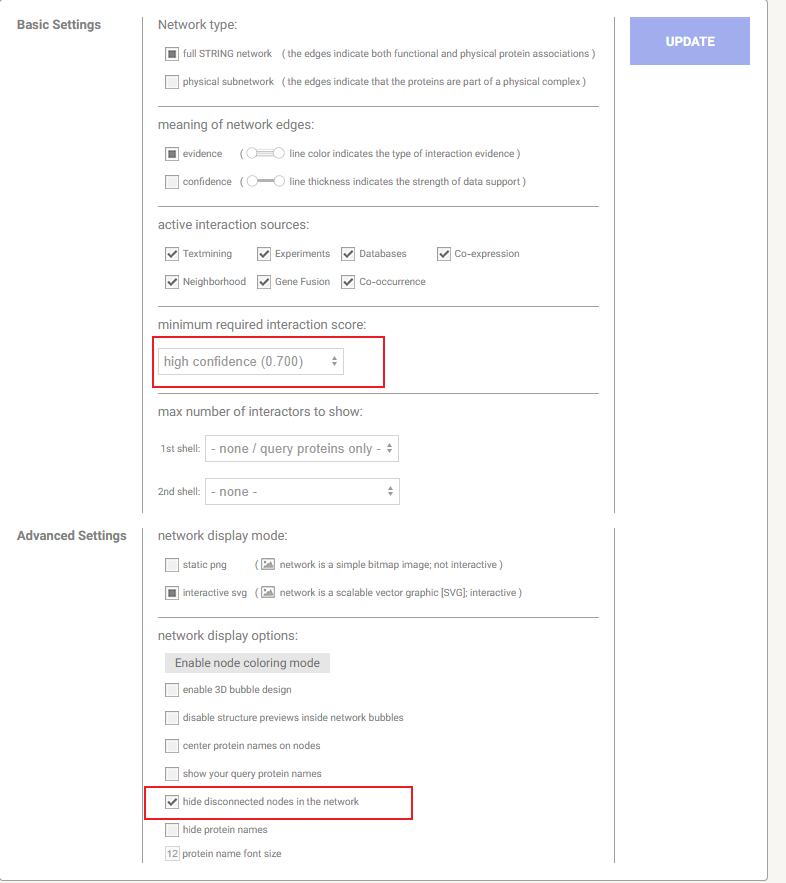
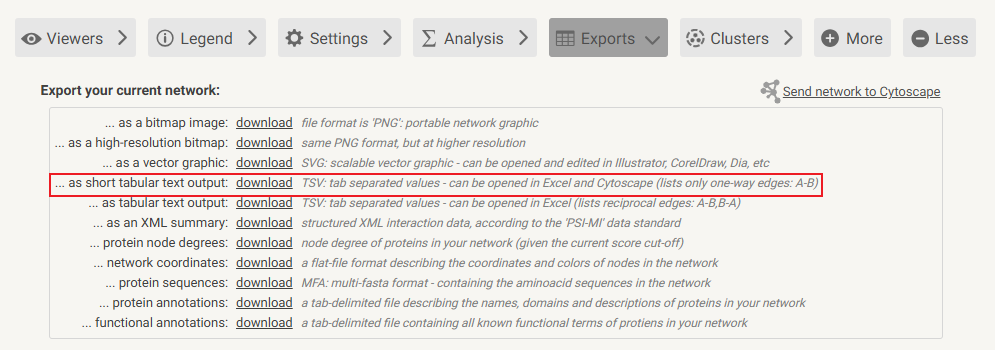
PPI
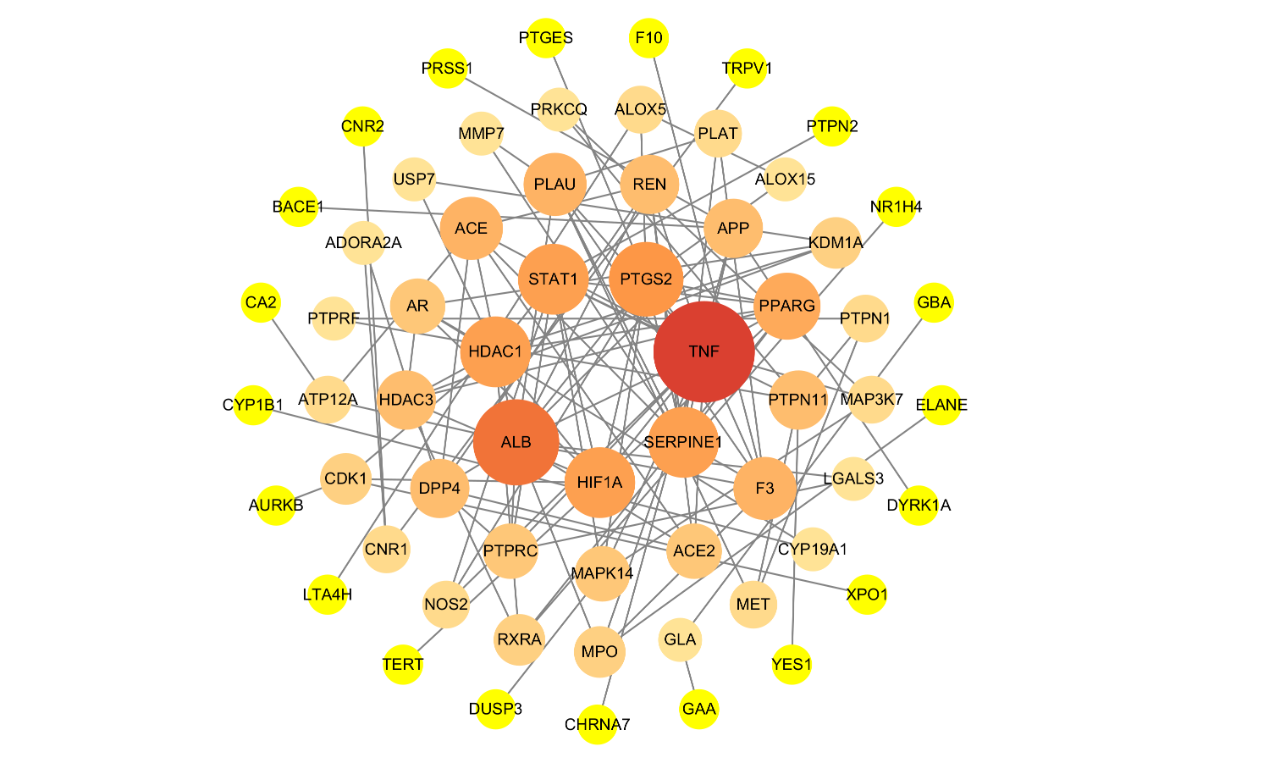
- # insert直接输出strings (选择人源)
成分-靶点-通路网络图
根据PPI网络图中展示的靶点,去找对应的成分,再找对应的通路,参考连钱草课题的流程
mol-gene-pathway
- # 将取交集得到的75个靶点输入 KEGG DAVID:DAVID: Functional Annotation Result Summary (ncifcrf.gov)。KEGG pathway富集分析得到通路,让Ghatgpt帮我选出了9个与糖尿病相关的通路,将其中的基因挑出来。
- # /home/yqyang/zzy/hsbd/np/mgp/mol-targets
- awk -F '"' '{print $2}' insert-kegg-pathway.csv|sed 's/, /\n/g'|sort|uniq >pathway-gene
- # id转换为名称
- for i in
`cat pathway-gene`;do grep -w "$i" ../../data/etcm-targets-yls.csv|awk -F ',' '{print $1}' >>gene-name.txt;done - # 根据名称检索对应的化合物
- while read -r i; do grep "$i" ../../data/etcm-mols-smi-targets-fyl.csv|awk -F ',' '{print $2}'>>pathway-gene-mols-smi; done < gene-name.txt
- # excel 里把挑中的那几行复制到新的表格中,保存为csv文件
- # 这个csv文件格式笃定:只有三列,第一列是序号,第二列是pathway,第三列是对应的基因
- # 在文件里把" 替换为"后接一个空格,数据更好看
- # /home/zjxu/zzy/lqc/mol-gene-pathway
- # 这一步想得到的是 gene-pathway的两列
- # 方便后续for循环传入变量
- sed -i 's/ /_/g' gcd-tp-kegg-10pathway.csv
- for i in
`awk -F ',' '{print $2}' gcd-tp-kegg-10pathway.csv|sed 's/ /_/g'`;do grep $i gcd-tp-kegg-10pathway.csv|awk -F '"' '{print $2}'|sed 's/,/\n/g'|sed "s/^/${i} /" >>test;done - # 写脚本,将上述思路流程化
- # 基于上一步得到的基因,找对应的成分
- # 最后,把基因的uniprot-id替换成名称
- # 匹配的时候,有两个问题:有部分id对应多个结果-取第一个;有一个id找不到“P0DP24 · CALM2_HUMAN”,在uniprot上查找,手动添加到d3carp-id_name.txt中
- # 对d3carp-id_name.txt 清洗
- for i in
`awk '{print $1}' d3carp-id_name.txt |sort|uniq`;do grep -w "$i" d3carp-id_name.txt|head -n1>>d3carp-id_name-uniq.txt;done - (base) [zjxu@DDB lqc]$ wc -l *txt
- 6111 d3carp-id_name.txt
- 6034 d3carp-id_name-uniq.txt