12.2 MD
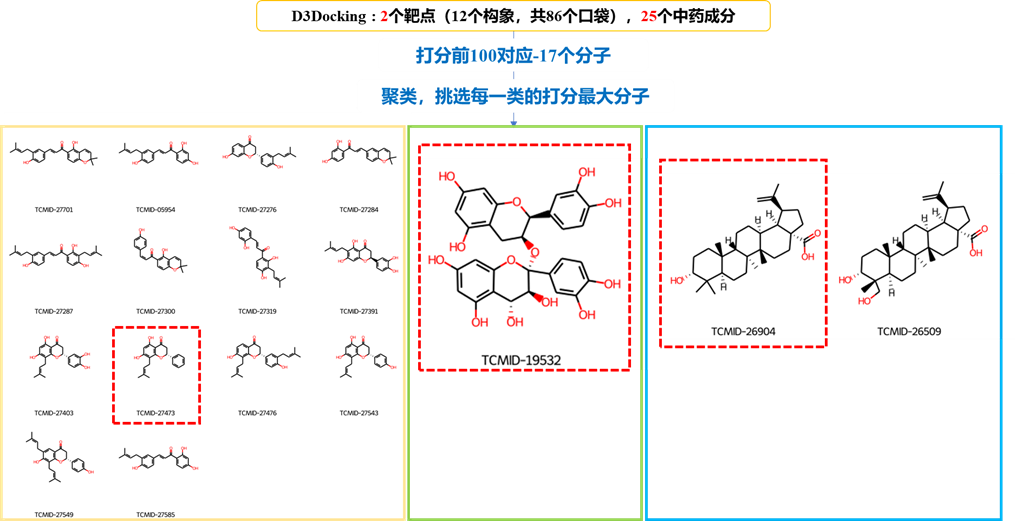
初始构象:D3Docking结果
- # 找到最高打分对应的复合物结构(靶点-口袋)
- # 172.21.85.12 /home/zjxu/zzy/hsbd/dock/1129/complex
- # 以 TCMID-27473 为例
- # ORF1ab_819-2763_Papain-like_proteinase+Dimer-TCMID-27473.txt 记录了每个口袋的打分
- # PDB文件夹里记录了对接原始结果,根据Pocket提取化合物的对接构象
- # 将3个体系的最好对接结果转移至 172.21.75.1
MD
生成配体参数
- # 172.21.75.1 /home/data/zzy/project/hsbd/lig
- 1. 获取三维结构(检查质子化情况!)
- D3Docking的输出结果是三维结构,但是没有非极性氢。用Schrodinger ligprep(本地2017),从输入的三维结构定义手性中心,pH=7.4。
- 2. 使用antechamber生成配体小分子参数(75.1)/home/data/zzy/project/hsbd/lig/g16
- # !!!!!!!!!!记得挨个检查电荷哇!!!!!!!
- # 26904 带一个负电(羧基),需要手动修改,再在gv里检查
- for i in
`cat index`;do test -e $i && rm -r $i;mkdir $i;cd $i;antechamber -i ../../2017maestro/${i}.sdf -fi sdf -o $i.gjf -fo gcrt;sed -i '1,3d' $i.gjf;sed -i '1i %mem=4GB\n%nproc=4\n#B3LYP/6-31G* Pop=MK iop(6/33=2) iop(6/42=6) opt' $i.gjf;cd ../;done
- # 4090上没找到g16,暂时到23上交任务
- # 172.21.85.23 /home/databank_70t/zzy/project/hsbd/g16/
- nohup g16 TCMID-19532.gjf &
- nohup g16 TCMID-26904.gjf &
- nohup g16 TCMID-27473.gjf &
- for i in
`cat index`;do cd $i;antechamber -i $i.log -fi gout -o $i.prep -fo prepi -c resp;parmchk2 -i $i.prep -f prepi -o $i.frcmod -a y;cd ../;done
蛋白准备
- # 蛋白质处理 /home/data/zzy/project/hsbd/pro
- # 检查了一下,D3Docking的结果里,蛋白是 质子化后的状态
- # 质子化脚本位置 172.21.85.12 /home/zjxu/ylshi/SARS-CoV-2-process
- # 尝试了一下,pdb2pqr处理这三个蛋白,两个会报错。tleap可以正常读取,遂不做多余处理,等待配体高斯优化完成后直接tleap
- # tleap 如果用D3Docking的原始输出Pdb 作为受体,会报错
- # 去85.12 上把对接的输入受体文件下载下来了(pdbqt),后缀改为pdb
- # 有缺失的序列!!!! 12.2 16:47
- # /home/data/zzy/project/hsbd/sys-pre/pro/pdb2pqr
- pdb2pqr30 ../raw/pdb/ORF1ab_3264-3569_3C-like_proteinase--Dimer.pdb 19532.pqr --ff AMBER --ffout AMBER --with-ph 7.4 --pdb-output 19532.pdb
- pdb2pqr30 ../raw/pdb/6w9c-Papain-like_proteinase_Trimer.pdb 26904.pqr --ff AMBER --ffout AMBER --with-ph 7.4 --pdb-output 26904.pdb
- pdb2pqr30 ../raw/pdb/ORF1ab_819-2763_Papain-like_proteinase--Dimer.pdb 27473.pqr --ff AMBER --ffout AMBER --with-ph 7.4 --pdb-output 27473.pdb
- # 手动删除非蛋白的行(非ATOM开头,通常是 HETATM 开头)
复合物体系准备
复合物初始构象的坐标就采用D3Docking的对接结果
gv-check
- # /home/data/zzy/project/hsbd/lig/gv
- for i in
`cat index`;do cp ../g16/TCMID-${i}/NEWPDB.PDB rawg16-${i}.pdb;done - for i in
`cat index`;do cp ../../complex-from-d3dock/TCMID-${i}/*sdf d3dock-${i}.sdf;done - # 本地 gv
- # gv里分别打开 NEWPDB.PDB(rawg16-${i}.pdb) 与 d3dock-${i}.sdf
- 打开GV tools-atom list-鼠标点击symbol列-rows-sort selected -拖动鼠标选择所有H-edit-delete-selected atoms),接着仍在atom list中,edit-z matrix-standardize,保存。
- 得到 g16-gv-${i}.pdb dock-${i}-gv.pdb
- # GV里 检查 g16-gv-${i}.pdb 与 dock-${i}-gv.pdb,更改 dock-${i}-gv.pdb (atom list - Tag),使其Tag 与g16-gv.pdb 完全一致,修改后得到 dock-${i}-gv-checked.pdb
- # /home/databank_70t/zzy/people/samuel/20241016-t13-5mol-cmd/sys-pre/gv-check
- dos2unix *
- sh gv-check-lig.sh
- #### "gv-check-lig.sh" 6L, 207C #############################################################
- for i in
`cat index` - do
- grep -v "CONECT" g16-gv-${i}.pdb |cut -b 1-26 > symbol-${i}
- grep -v "CONECT" dock-${i}-gv-checked.pdb | cut -b 29-78 > coordinate-${i}
- paste -d " " symbol-${i} coordinate-${i} > lig-${i}.pdb
- done
- #############################################################################################
tleap
- # 172.21.75.1 /home/data/zzy/project/hsbd/sys-pre/tleap
- # sh tleap.sh
- # 检查复合物结构是否合理
- # /home/data/zzy/project/hsbd/sys-pre/tleap/check
- for i in
`cat ../../index`;do echo 18|gmx_mpi trjconv -s ../$i/gmx/gromacs.gro -f ../$i/gmx/gromacs.gro -n ../$i/gmx/index.ndx -o $i.pdb;done - # 与对接结果 /home/data/zzy/project/hsbd/sys-pre/complex-from-d3dock 进行对比
- # 唯一需要外部引入的参数是抗衡离子数量( vi tleap.in)
- # 第一次运行后,去leap.log里看一下,蛋白的带电情况(配体都是电中性)
- # 本次实验中,3个体系蛋白不同,且都是多聚体,需要额外检查top文件与posre.itp文件
体系平衡
- # 172.21.75.1 /home/data/zzy/project/hsbd/pre-bal
- scp -r dddc@172.21.85.2:/home/dddc/zzy/project/trip13/20241016-t13-5mol-cmd/pre-equilibrium/mdp ./
- nohup sh min-nvt-npt.sh &
200ns模拟记录
19532
- # 172.21.75.1 /home/data/zzy/project/hsbd/200ns-cmd/19532/
- source ~/.gmx-2022.5-GPU.sh
- dddc@gpu-4090:/home/data/zzy/project/hsbd/200ns-cmd/19532/1$ nohup gmx_mpi mdrun -v -deffnm md &
- [1] 3339463
- export CUDA_VISIBLE_DEVICES=1
- dddc@gpu-4090:/home/data/zzy/project/hsbd/200ns-cmd/19532/2$ nohup gmx_mpi mdrun -v -deffnm md &
- [2] 3340079
- export CUDA_VISIBLE_DEVICES=2
- dddc@gpu-4090:/home/data/zzy/project/hsbd/200ns-cmd/19532/3$ nohup gmx_mpi mdrun -v -deffnm md &
- [3] 3340188
26904
- # 172.21.85.9 /home/databank/zzy/project/hsbd/200ns-cmd/26904
- scp -r dddc@172.21.75.1:/home/data/zzy/project/hsbd/200ns-cmd ./
- source ~/.gmx2022.5-remd.sh (export CUDA_VISIBLE_DEVICES=2)
- (base) [chpeng@localhost 1]$ nohup gmx_mpi mdrun -v -deffnm md &
- [1] 61965
- export CUDA_VISIBLE_DEVICES=0
- (base) [chpeng@localhost 2]$ nohup gmx_mpi mdrun -v -deffnm md &
- [2] 61993
- export CUDA_VISIBLE_DEVICES=1
- (base) [chpeng@localhost 3]$ nohup gmx_mpi mdrun -v -deffnm md &
- [3] 62013

