4.1 阳性化合物MD
目前文章中,5个体系的结合位点仅有一个体系是有过报道的,这样没什么可说的。
与徐老师讨论后,希望再补充天然产物在已知位点的MD,都使用阳性化合物的结合位点,对接我们的化合物,作为初始构象去跑CMD,看看与D3Docking的结果相比,哪个结合自由能更好。
这样讨论起来,更充分些。也有助于进一步说明原子层面上的相互作用
另一方面,跟徐老师提了一下5个活性成分的入血问题,给徐老师提供了成分信息,去问许海玉老师,他们是否被确证为HSBD的活性成分。
蛋白准备
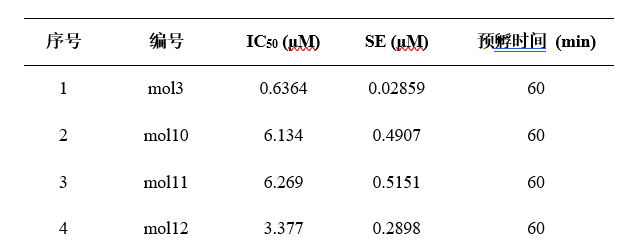
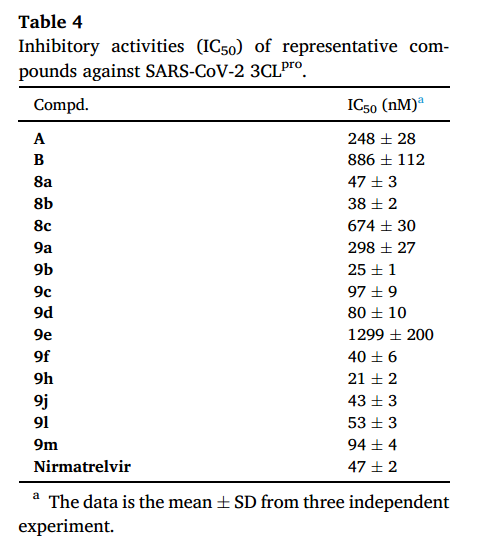
PL的阳性对照为GRL0617,复合物晶体结构 PDB:7JRN (分辨率最好)
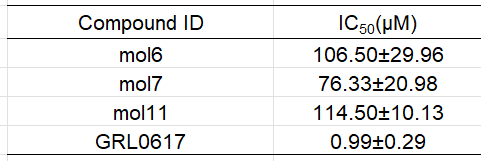
3CL的阳性对照为Nirmatrelvir,复合物晶体结构 PDB:7VH8 (EJMC文章中用的结构)
体系离子检查
PLpro中,晶体结构中有硫酸根离子/锌离子
徐老师的建议:
- 硫酸根离子一般是晶体结构解析所用的,MD不需要考虑
- 锌离子如果和配体存在碰撞那也是要删除的,一般结合位点的锌离子会起到稳定负电荷的作用,但是目前这个体系中不存在这种情况,可删除
二硫键检查
- pymol -show- Disulfide bonds(无)
- pymol - select resn cys -show sphere 看硫原子距离,一般需要在范德华半径之内 (无)
Pymol自带的判断还是比较准确的
蛋白准备(晶体结构)
- 85.23 /home/databank_70t/zzy/project/hsbd/active_control/sys-pre/pro/pdb2pqr
- pdb2pqr30 ../7jrn.pdb 7jrn.pqr --ff AMBER --ffout AMBER --with-ph 7.4 --pdb-output 7jrn_complex.pdb
- grep -v HETATM 7jrn_complex.pdb |awk '$5=="A"{print}' >7jrn_apo_clean.pdb
- pdb2pqr30 ../7vh8.pdb 7vh8.pqr --ff AMBER --ffout AMBER --with-ph 7.4 --pdb-output 7vh8_complex.pdb
- grep -v HETATM 7vh8_complex.pdb >7vh8_apo_clean.pdb
配体准备
坐标信息(dock)
- 23 /home/databank_70t/zzy/project/hsbd/known-plt/3cl/sys-pre/ligand/dock
- for i in
`cat index`;do smina --seed 1 --num_modes 10 -r ../../pro/pdb2pqr/7vh8_apo_clean.pdb -l ../ligprep/${i}.sdf --autobox_ligand 7vh8_B_4WI.sdf -o smina_${i}.sdf;done - 23 /home/databank_70t/zzy/project/hsbd/known-plt/pl/sys-pre/ligand/dock
- for i in
`cat index`;do smina --seed 1 --num_modes 10 -r ../../pro/pdb2pqr/7jrn_apo_clean.pdb -l ../ligprep/${i}.sdf --autobox_ligand 7jrn_C_TTT.sdf -o smina_${i}.sdf;done
质子化预测
- 23 /home/databank_70t/zzy/project/hsbd/known-plt/3cl/sys-pre/ligand/ligprep
- 23 /home/databank_70t/zzy/project/hsbd/known-plt/pl/sys-pre/ligand/ligprep
- cp ../ligprep/index ./
- # 检查输入文件的位置(do.sh)
- sh do.sh
高斯优化
- 23 /home/databank_70t/zzy/project/hsbd/known-plt/3cl/sys-pre/ligand/g16
- 23 /home/databank_70t/zzy/project/hsbd/known-plt/pl/sys-pre/ligand/g16
- # 使用antechamber生成配体小分子参数(85.23)
- for i in
`cat index`;do mkdir $i;cd $i;antechamber -i ../../ligprep/${i}.sdf -fi sdf -o $i.gjf -fo gcrt;sed -i '1,3d' $i.gjf;sed -i '1i %mem=4GB\n%nproc=4\n#B3LYP/6-31G* Pop=MK iop(6/33=2) iop(6/42=6) opt' $i.gjf;cd ../;done - # 提交 高斯优化任务 15:45
- # xx:xx 完成
- for i in
`cat index`;do cd $i;antechamber -i $i.log -fi gout -o $i.prep -fo prepi -c resp;parmchk2 -i $i.prep -f prepi -o $i.frcmod -a y;cd ../;done
atom_name_check
- 23 /home/databank_70t/zzy/project/hsbd/known-plt/3cl/sys-pre/ligand/atom_name_check
- 23 /home/databank_70t/zzy/project/hsbd/known-plt/pl/sys-pre/ligand/atom_name_check
- sh do.sh
- # 可视化检查
tleap
- # 加载环境
- source ~/zzy/ff19sb.sh
- # 23 /home/databank_70t/zzy/project/hsbd/known-plt/3cl/sys-pre/tleap
- for i in
`cat index`;do mkdir -p $i;cp ../ligand/g16/${i}/${i}.frcmod ./${i}/lig.frcmod;cp ../ligand/g16/${i}/${i}.prep ./${i}/lig.prep;cp ../ligand/atom_name_check/${i}/LIG.PDB ./${i}/lig.pdb;cp ../pro/pdb2pqr/7vh8_apo_clean.pdb ./${i}/pro.pdb;cp md_parm_gen_chinese.py $i;cd $i;python md_parm_gen_chinese.py 1;cd ../;done - # 23 /home/databank_70t/zzy/project/hsbd/known-plt/pl/sys-pre/tleap
- for i in
`cat index`;do mkdir -p $i;cp ../ligand/g16/${i}/${i}.frcmod ./${i}/lig.frcmod;cp ../ligand/g16/${i}/${i}.prep ./${i}/lig.prep;cp ../ligand/atom_name_check/${i}/LIG.PDB ./${i}/lig.pdb;cp ../pro/pdb2pqr/7jrn_apo_clean.pdb ./${i}/pro.pdb;cp md_parm_gen_chinese.py $i;cd $i;python md_parm_gen_chinese.py 1;cd ../;done - # 可视化检查
amber_to_gmx
- # 23 /home/databank_70t/zzy/project/hsbd/known-plt/3cl/sys-pre/amber_to_gmx
- bash do.sh
- # 根据配体原子数生成 posre2.itp (原子数+4=行数)
- sed -n '1,46p' 56083-03-5-dock-3CL/posre1.itp > 56083-03-5-dock-3CL/posre2.itp
- sed -n '1,61p' 59870-65-4-dock-3CL/posre1.itp > 59870-65-4-dock-3CL/posre2.itp
- # 23 /home/databank_70t/zzy/project/hsbd/known-plt/pl/sys-pre/amber_to_gmx
- sed -n '1,39p' 1486-70-0-dock-PL/posre1.itp > 1486-70-0-dock-PL/posre2.itp
- sed -n '1,46p' 56083-03-5-dock-PL/posre1.itp > 56083-03-5-dock-PL/posre2.itp
- sed -n '1,59p' 91433-17-9-dock-PL/posre1.itp > 91433-17-9-dock-PL/posre2.itp
预平衡
- # 8-4090(75.3)/home/dddc/zyzhou/project/hsbd/known-pkt/pl/pre-equ
- cp ../sys-pre/amber_to_gmx/index ./
- cp -r ../../3cl/pre-equ/mdp/ ./
- cp -r ../../3cl/pre-equ/pre_equ.py ./
- for i in
`cat index`;do mkdir -p $i/pre-equ;cp -r ../sys-pre/amber_to_gmx/$i $i/parameters;cp -r mdp/ $i/;done - # xcy脚本出现问题,4-2进行系统测试,另附详情
- # 暂且使用zzy之前的预平衡方法及mdp
- # em.mdp是否需要位置限制,限制哪些原子,需要深思
MD模拟任务
- # 见 75.3 /home/dddc/zyzhou/project/hsbd/ 22:53
- # 提交2个体系的3次平行模拟
