0918 SMD新流程
概述
- # CV设置为关键氨基酸的主链
- # 对蛋白主链施加位置限制
- 85.24 /home/databank/zzy/project/MD/koff/smd/cv-res_bb-mol-restbb-0918
CV
- # trypsin 体系 (!!backbone 没有氧原子!!)
- # Hiv 体系 (!!backbone 没有氧原子!!)
SMD
- # 生成 index
- gmx_mpi make_ndx -f params/npt.gro -o index.ndx
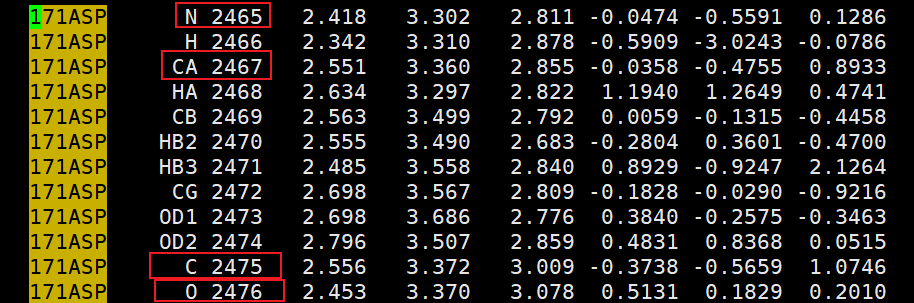
- > a2465|a2467|a2475 (trypsin)
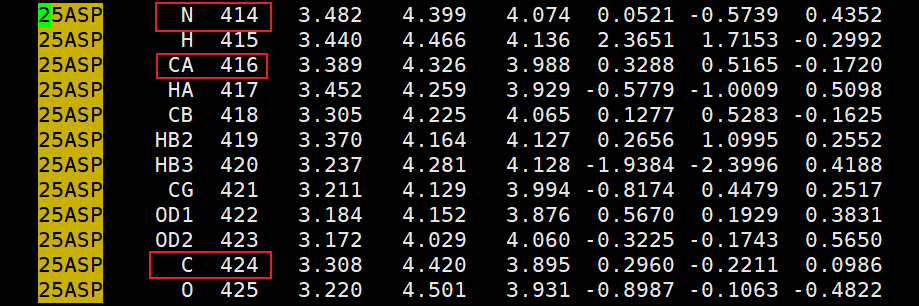
- > a414|a416|a424|a1983|a1985|a1993 (hiv)
- > protein|MOL
- # 生成 itp,规定被位置限制的原子
- gmx_mpi genrestr -f npt.gro -o rest-bb.itp
- Select a group: 4
- Selected 4: 'Backbone'
- # 在gmx.top 中删掉其他itp链接,更改蛋白后面的那个itp名字
- # tpr
- gmx_mpi grompp -f smd.mdp -p ../params/gmx.top -c ../params/npt.gro -r ../params/npt.gro -n ../params/index.ndx -o smd.tpr -maxwarn 1
- nohup mpirun -np 100 gmx_mpi mdrun -v -deffnm smd &
- # vis 检查轨迹
- gmx_mpi trjconv -f ../smd.xtc -s ../smd.tpr -n ../../params/index.ndx -o dry_traj.pdb -pbc mol -ur compact
- sort -g -k2nr ../smd_pullf.xvg |head
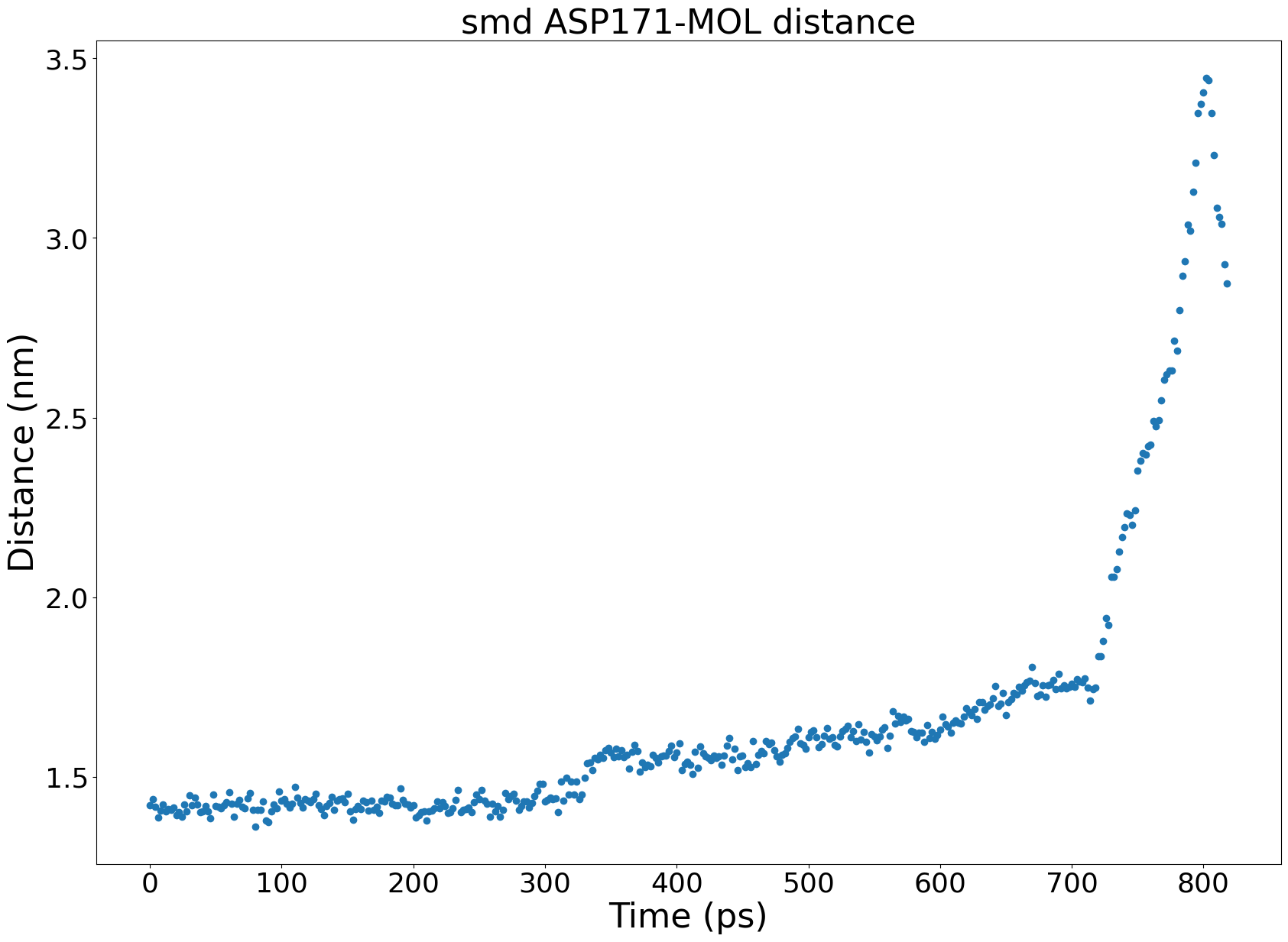
- # cpptraj rms 一下,跟 dry.pdb 比较关键氨基酸位置差异
fig
- # 85.24
- # /home/databank/zzy/project/MD/koff/trypsin/complex-pre/trypsin-31/gmx-smd/vis/test1/0918
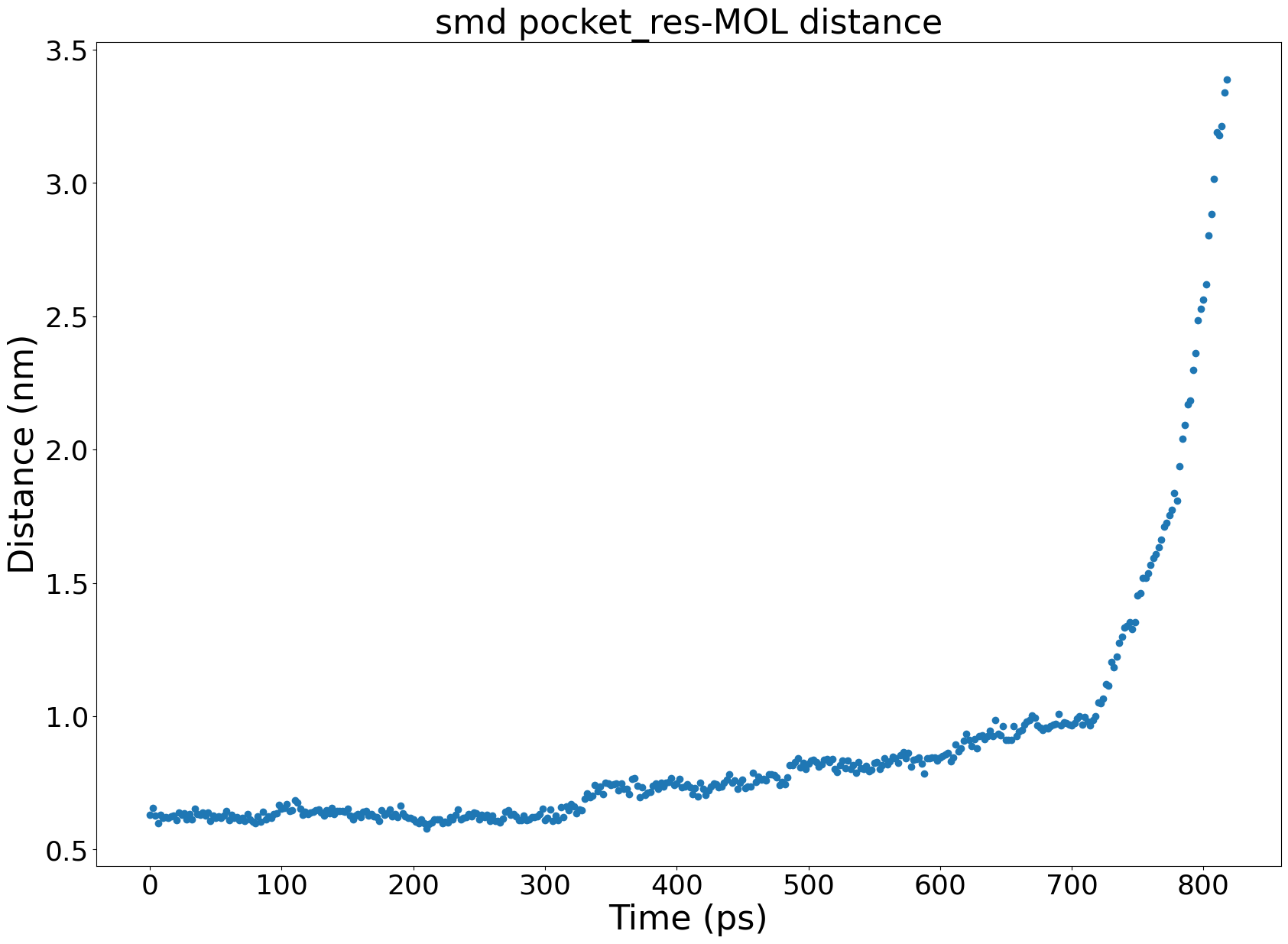
- # smd 的设置里其实是 固定了速度的,但是轨迹看下来并不是恒速,画个图确认一下
- # 这一轮测试的cv是口袋残基到配体的距离
- # 首先是基于smd自己产生的smd_pullx.xvg 得到的图是 smd pull x (ori)
- # 再用gmx计算下dis
- gmx_mpi distance -f dry.xtc -s ../dry.pdb -n ../../index.ndx -select 'com of resnr 171 172 173 174 177 197 198 196 204 195 205 194 206 193 192 40 81 79 78 plus com of resid 225' -oall dis-pocket-MOL.xvg
- # 可以看到中间有些点是缺失的,直观上的感觉,gmx-smd设置的速度太快了?