2.19 HIV 5sys align to crystal
SMD 初始构象获取
只有两个体系有初始结构
两个思路:
- 根据复合物初始结构,将其他5个体系的配体align上去
- 将配体对接
薛定谔的flexible alignment 只考虑了配体,叠合后配体会与蛋白有碰撞
Hermite FEP 在构建微扰图之前,可以进行constraint dock,从之前的经验看符合要求,进行尝试
上传蛋白(apo)
上传配体
晶体结构配体只提供坐标信息,pymol里另存为sdf格式即可(tleap原本是pdb格式)
5个体系的配体使用高斯优化-antechamber得到的NEWPDB.PDB,obabel转为sdf
- 85.23 /home/databank_70t/zzy/project/koff/smd/hiv-1/sys-pre/ligand/g16/newbpdb/sdf
- for i in
`cat index`;do obabel -ipdb ../${i}-NEWPDB.pdb -osdf -O ${i}-NEWPDB.sdf;done
提交 Constrained Dock 任务
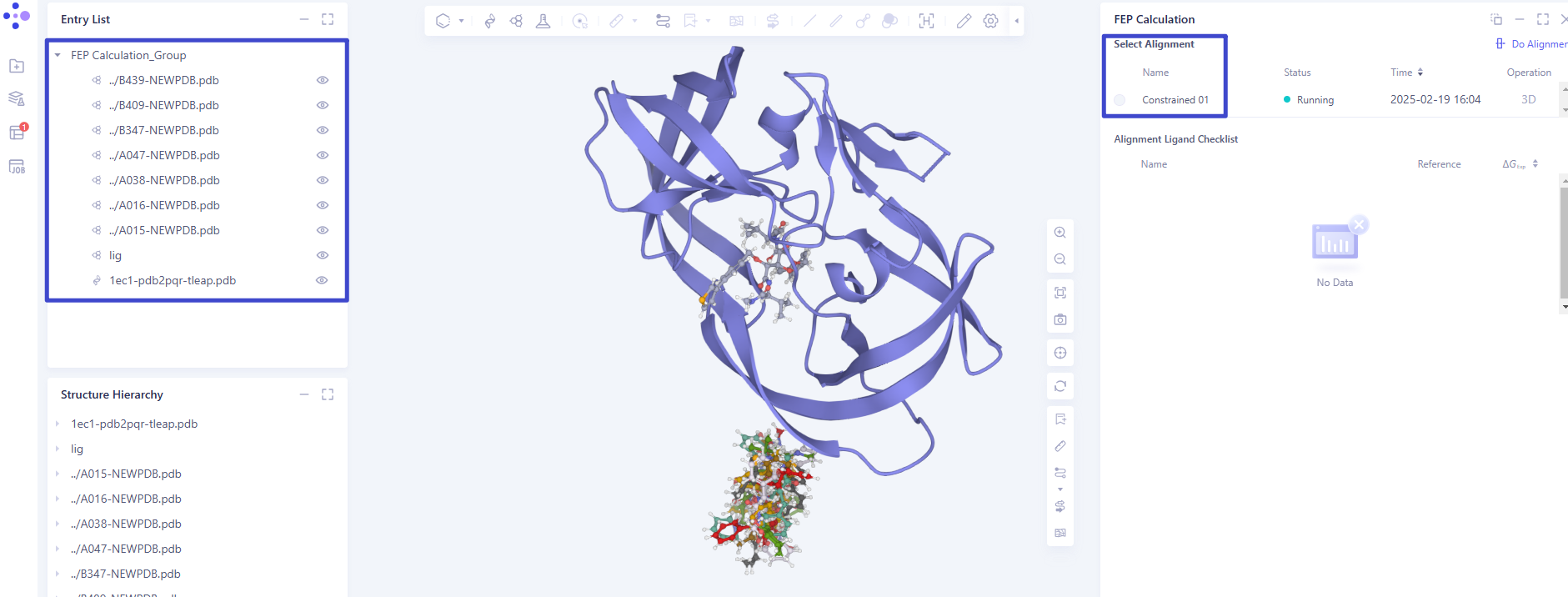
16:05 任务已提交 等待中
任务完成后,文件无法直接下载,只能进行ligand prepararion(所有参数都不选中),retain conformation能保持原有坐标,进行下载
- 23 /home/databank_70t/zzy/project/koff/smd/hiv-1/sys-pre/complex/5sys-hermite-constrained_dock
gv-check
- # 本地 gv
- # "HIV 5sys dock CMD"中已经得到了 g16-gv-${i}.pdb
- # 直接将${i}-hermite-CD.sdf 经过去氢,z matrix-standardize,check
- # 发现z matrix-standardize和index基本一致,神奇
- # 仅有 A038-hermite-CD-nc.pdb 需要检查(nc 意为need check)
- # 直接保存为 ${i}-hermite-CD.pdb
- # 23 /home/databank_70t/zzy/project/koff/smd/hiv-1/sys-pre/ligand/gv-check/hermite-CD
- dos2unix *
- sh gv-check-lig.sh
- #### "gv-check-lig.sh" 6L, 207C #############################################################
- for i in
`cat index` - do
- grep -v "CONECT" g16-gv-${i}.pdb |cut -b 1-26 > symbol-${i}
- grep -v "CONECT" ${i}-hermite-CD.pdb | sed '1d'|cut -b 29-78 > coordinate-${i}
- paste -d " " symbol-${i} coordinate-${i} > lig-${i}.pdb
- rm symbol-${i} coordinate-${i}
- done
- #####################################################################################
tleap
- # 23 /home/databank_70t/zzy/project/koff/smd/hiv-1/sys-pre/tleap2
- # 加载环境
- source ~/zzy/ff19sb.sh
- # 自动化流程 tleap
- nohup sh tleap.sh
- # 参数与"HIV 5sys dock CMD"一致(本质上只是配体的坐标信息不同)
- # 该体系是同源二聚体,posre1.itp 注意要手动修改(1-1569)
- # 这个修改在下一步执行的服务器上进行(85.9)
- 85.9 /home/databank/zzy/project/smd/hiv-1/sys-pre/tleap2
- cp -r ../tleap/posre1/ ./
- rm */gmx/posre1.itp
- for i in
`cat index`;do cp posre1/posre1.itp ./$i/gmx/;done
pre-equilibrium
- 85.9 /home/data/zzy/koff/smd/hiv-1/pre-equilibrium2
- source ~/zzy/gmx2024.4-gpu.sh
- (base) [chpeng@localhost pre-equilibrium2]$ nohup bash min-nvt-npt.sh &
- [1] 55047
- # 另外两个体系之前预平衡已完成,直接复制过来
- cp -r ../pre-equilibrium/A047/ ./
- cp -r ../pre-equilibrium/B409/ .
输入文件
- 85.9 /home/databank/zzy/project/smd/hiv-1/smd2/
- for i in
`cat index`;do mkdir -p $i;cp ../../sys-pre/tleap2/$i/gmx/topol.top ./$i/gmx.top;done - # 本地可视化检查npt.gro
- # 删除 top 文件中 posre2.itp 对应的那几行,posre1改为posre
- for i in
`cat index`;do vi $i/*top;done - for i in
`cat index`;do sed -i 's/posre1/posre/g' $i/gmx.top;done - for i in
`cat index`;do cp ../../pre-equilibrium2/$i/npt/*gro ./$i/npt.gro;done
CV
- # 85.9 /home/databank/zzy/project/smd/hiv-1/smd2/cv
- cp -r ../7sys-npt_top/* ./
- ####################"get_pkt_3A_atom_index.sh" 8L, 185C #############################
- for i in
`cat index` - do
- cd $i
- cpptraj -i ../mask_MOL_3A.in
- awk '{print $2}' mol-3A.dat |sed '1d'|sed ":a;N;s/\n/|a/g;ta" |sed 's/^/a/'|sed 's/|a$//' >> ../pkt_3A_atom_index
- cd ..
- done
- ######################### "mask_MOL_3A.in" 4L, 119C ###############################
- parm gmx.top
- trajin npt.gro
- mask "(:199<:3.0)&((!:WAT)&(!:MOL))&(@N,CA,C,O)" maskout mol-3A.dat maskpdb mol-3A.pdb
- run
- #####################################################################################
- sh get_pkt_3A_atom_index.sh
- # 得到 pkt_3A_atom_index 包含了每个体系中配体周围3A的原子index,用于设置pull group
SMD-umbrella
- # 85.9 /home/databank/zzy/project/smd/hiv-1/smd2/umbrella
- source ~/zzy/gmx2024.4-gpu.sh
- export OMP_NUM_THREADS=20
- # posre.itp 得检查,1569后的原子序号top中不存在,得删去
- # index.ndx 中的group名称长度有上限,因此在Index.ndx中将"a_132_a_133_a_134_a_135_a_136_a_137 ...." 这样一大串替换为“pkt_3A_atom_index”
- mdp文件里也同步替换pull_group1_name
- # 需要设置pbc参考原子,尝试用之前B409体系的pull group CG 1990
- pull-group1-pbcatom = 1990
- cp ../../smd/umbrella/* ./
- paste ../cv/index ../cv/pkt_3A_atom_index >index
- cp -r ../7sys-npt_top/* ./
- nohup bash smd.sh &
数据分析
- # 85.9 /home/databank/zzy/project/smd/hiv-1/smd2/umbrella/analysis
- bash smd-ana.sh
