轨迹处理&分析
周期性处理
gmx -pbc mol -ur compact (-s 接tpr)(配位离子需要保留,保留溶质组)(保留输出轨迹) output file 命名:dry.xtc
cpptraj autoimage (parm prmtop)(保留输出轨迹) output file 命名:dry-autoimage.xtc
RMSD
大致检查周期性处理情况
全原子叠合
全原子计算
gmx rms (算溶质的rms)
apo/complex 口袋RMSD
ref 为complex的初始结构(crystal)口袋氨基酸
rmsd 计算 使用 backbone 来叠合(fit)
计算 rmsd值 仅考虑重原子
看apo 1*3 um 内是否出现 与complex 接近的构象(rmsd接近0 的 构象占比)
看complex 的 rmsd曲线
关键差异氨基酸RMSF
按D3PM 库中标注的最大差异氨基酸为准
先按照配体,选择周围(即口袋)氨基酸计算RMSF
半柔性对接中蛋白构象对晶体结构复现的影响
期待结果:
- apo晶体结构无法复现配体结合构象
- 蛋白构象(口袋的正确结构)引起的对接打分差异
测试
- 75.2 /home/databank/MD_traj_dataset/D3PM/1um_cmd_12A/batch2
- # batch1 3vvx_B_pair4 3vw0_D_pair4 没跑完,正在转移至75.3 ,转移完成后续跑
检查轨迹的完整性
- # 1um是否跑完了
- for a in *;do for i in {1..3};do echo "########## $a md$i ##########";grep -A 2 Step ${a}/md${i}/*log|tail -n5;done;done
- # 轨迹文件是否完整
- for a in *;do du -sh ${a}/*;done
pbc
bash
- # batch2的pbc任务均已提交
- 75.2 /home/databank/MD_traj_dataset/D3PM/analysis/pbc/complex
- 75.2 /home/databank/MD_traj_dataset/D3PM/analysis/pbc/apo
- # 已撰写好了流程化脚本,后面按照这种方法进行周期性处理,脚本已测试通过(截取了很短的轨迹做测试)
- 75.2 /home/databank/MD_traj_dataset/D3PM/analysis/pbc2_test
- # 0910 脚本已经开始运行
- 75.2 /home/databank/MD_traj_dataset/D3PM/analysis/batch3/pbc
- # 处理不同批次下的体系,需要更改 pbc.sh 里的 datadir 参数(用于读取tpr和原始轨迹)
- # 同时,需要更改 autoimage-apo.sh 和 autoimage-complex.sh 的参数读取路径
- # cpptraj 所需参数需要从v100服务器复制
- # 以batch3为例,路径为
- 75.2 /home/databank/MD_traj_dataset/D3PM/analysis/batch3/sys_md_parameters
- # 0910 因为目前75.2上只有4对体系可进行处理,参数只打包了这4对
python
2025-09-10 16:35 版本如上,测试路径如下:
- 75.2 /home/databank/MD_traj_dataset/D3PM/analysis/batch3/pbc2
其中 autoimage 是外接bash 脚本实现的,pytraj autoimage 在测试中报错,找不到盒子信息,加上其读取轨迹速度慢,暂不使用pytraj autoimage。
rmsd的计算 是使用rmsd,按照下午和师姐的讨论结果,apo和complex都是使用复合物的晶体构象作为参考构象计算
autoimage
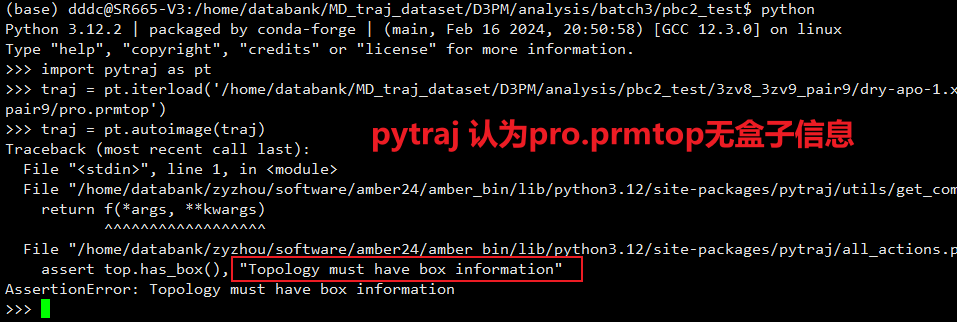
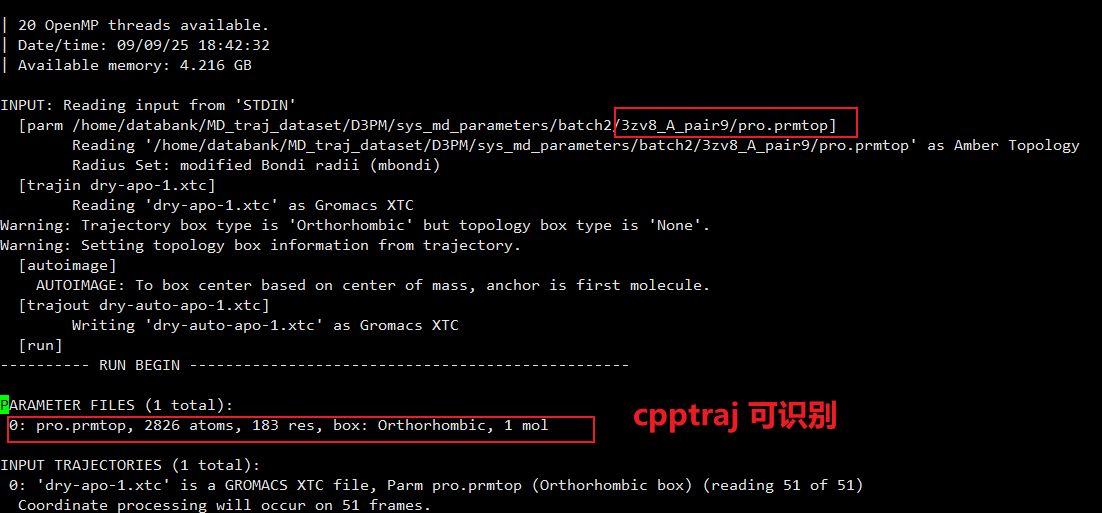
RMSD
师姐的建议
- import os
- import pandas as pd
- import numpy as np
- import pytraj as pt
- #trajectory of 0-500ns
- traj = pt.iterload('/home/databank/lywu/apo-proteins/ache/analysis/xtc/0_fit.xtc',top='/home/databank/lywu/apo-proteins/ache/analysis/xtc/dry.pdb')
- rmsf_data = pt.rmsf(traj,options='byres')
- df=pd.DataFrame(rmsf_data)
- if not os.path.exists("csv_rmsf"):
- os.makedirs("csv_rmsf")
- df.to_csv('./csv_rmsf/rmsf_apo_ache-md0_0-500ns.csv', index=False)
- import os
- import pandas as pd
- import numpy as np
- import pytraj as pt
- traj = pt.iterload('/home/databank/MD_traj_dataset/D3PM/analysis/batch3/pbc2_test/4E0F_pair18_pair18/dry-auto-apo-1.xtc',top='/home/databank/MD_traj_dataset/D3PM/analysis/batch3/sys_md_parameters/4E0F_B_pair18/pro.prmtop')
- rmsd_data = pt.rmsd(traj)
- df=pd.DataFrame(rmsd_data)
- if not os.path.exists("csv_rmsd"):
- os.makedirs("csv_rmsd")
- df.to_csv('./csv_rmsd/test_apo_rmsd.csv', index=False)
pkt-rmsd
- 75.2 /home/databank/MD_traj_dataset/D3PM/analysis/pkt_rmsd
- # 脚本已初步测试通过,可以计算 口袋rmsd
- # 初步检查 apo 和 complex 轨迹前几帧的rmsd,理论上,因为参考构象是complex初始结构,complex应该是0。测试得到的结果显示,complex接近0,apo不是0(如下图)

