HSP90 alpha vsREMD
根据1月2号的讨论,做两个体系:一个是heliex 的apo,一个是 loop 的apo,副本的初始构象之间无差异。放在服务器上跑500ns,看最后能不能收敛。
初始结构选择
在UniProt上查找 P07900 · HS90A_HUMAN(732aa ),把structure 数据复制到exce表里,用于根据分辨率排序并看氨基酸序列位置(大多是1-239aa这个区间,有不少缺失片段)
PDB 上限定关键词 apo P07900 一共就只有6个结果。其他的几百个结构都是复合物,还挺有意思。
明天过来,把几个结构 align 到一起看一下loop 还是heliex。
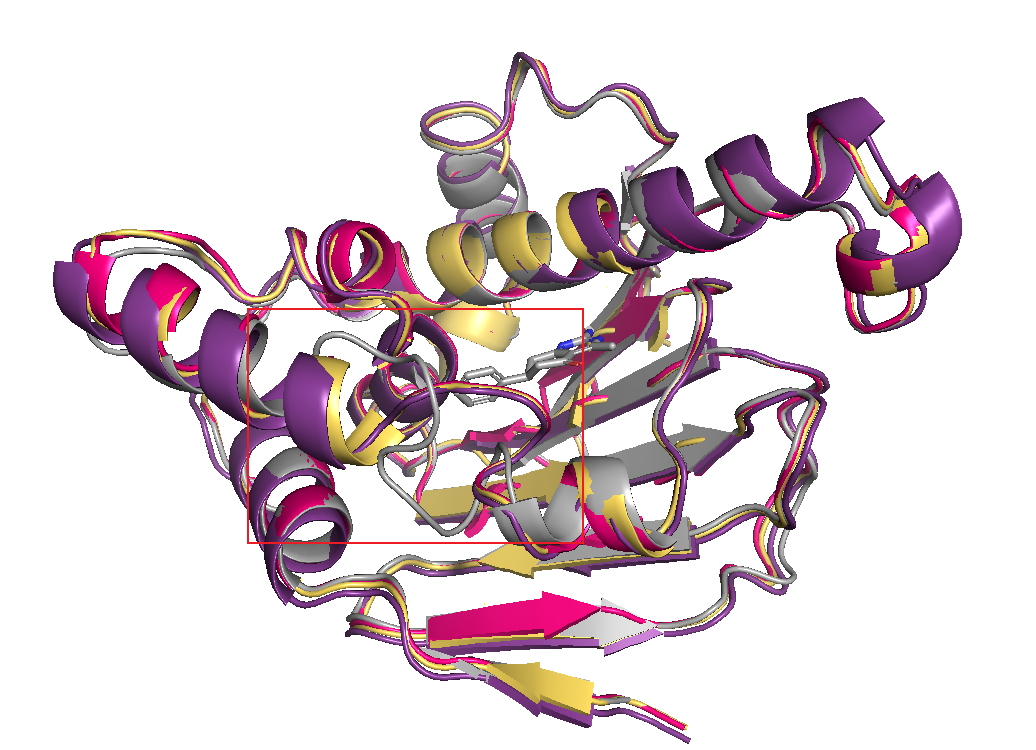
灰色的 (pdb id: 4w7t) 的构象是第三种-更大的loop区域
- 85.24 /home/databank/zzy/project/MD/koff/smd/HSP90/apo-vsremd
- # 5J80是目前分辨率最好的构象,口袋区域的heliex/loop区域是 loop
- # 根据分辨率从好到差寻找 binding site 是 heliex的构象,前8个都是loop,直到第9个才是heliex
- # 跟罗姐交流:hsp90 alpha 有三种构象,loop分为open/close两种,上面说的(pdb id: 4w7t) 的构象即是open的,与其相对,大部分都是close的loop
初始构象处理
- # PDB 上下载 PDB ID:5J80
- # 与师姐讨论序列长度的问题:师姐建议去查找文献中对于该domain的定义
序列长度是1-236,需要找koff实验中用的那种蛋白(如果实验测的是突变体,就用突变体),补链的问题,目前的晶体结构中1-15,225-236基本都是missing,AF2的结构很不合理且会使盒子大小变大很多,需要组会上与老师们讨论。
- # pdb2pqr
- pdb2pqr30 ../7s9h.pdb 7s9h.pqr --ff AMBER --ffout AMBER --with-ph 7.4 --drop-water --pdb-output 7s9h-pdb2pqr.pdb
